library(ggplot2)
library(betareg)
library(cmdstanr)
## This is cmdstanr version 0.8.1
## - CmdStanR documentation and vignettes: mc-stan.org/cmdstanr
## - CmdStan path: /usr/local/cmdstan
## - CmdStan version: 2.36.0
library(posterior)
## This is posterior version 1.6.1
##
## 次のパッケージを付け加えます: 'posterior'
## 以下のオブジェクトは 'package:stats' からマスクされています:
##
## mad, sd, var
## 以下のオブジェクトは 'package:base' からマスクされています:
##
## %in%, match
library(bayesplot)
## This is bayesplot version 1.11.1
## - Online documentation and vignettes at mc-stan.org/bayesplot
## - bayesplot theme set to bayesplot::theme_default()
## * Does _not_ affect other ggplot2 plots
## * See ?bayesplot_theme_set for details on theme setting
##
## 次のパッケージを付け加えます: 'bayesplot'
## 以下のオブジェクトは 'package:posterior' からマスクされています:
##
## rhat
knitr::knit_engines$set(stan = cmdstanr::eng_cmdstan)(0, 1)の範囲をとるデータに対する回帰にはベータ回帰がよく使われます。Rのbetaregパッケージを使った場合とStanを使った場合、さらに単純に線形回帰を当てはめた場合を比較してみました。
準備
データ生成
以下のモデルで、ベータ分布に従うデータ\(Y\)を生成します。平均\(\mu\)をパラメータとして使う関係で、パラメータ\(\kappa\)も設定します。
\[ Y \sim \mathrm{Beta}(\alpha, \beta) \\ \mu = \frac{\alpha}{\alpha + \beta} = \mathrm{logit}^{-1}(-4.5 + 0.5 X) \\ \kappa = \alpha + \beta \]
set.seed(1234)
inv_logit <- function(x) {
1 / (1 + exp(-x))
}
N <- 100
X <- runif(N, 0, 10)
logit_mu <- -4.5 + 0.7 * X
mu <-inv_logit(logit_mu)
kappa <- 6
alpha <- mu * kappa
beta <- (1 - mu) * kappa
Y <- rbeta(N, alpha, beta)
sim_data <- data.frame(X = X, Y = Y)データ確認
データを図示します。
p0 <- ggplot(sim_data, aes(x = X, y = Y)) +
geom_point()
print(p0)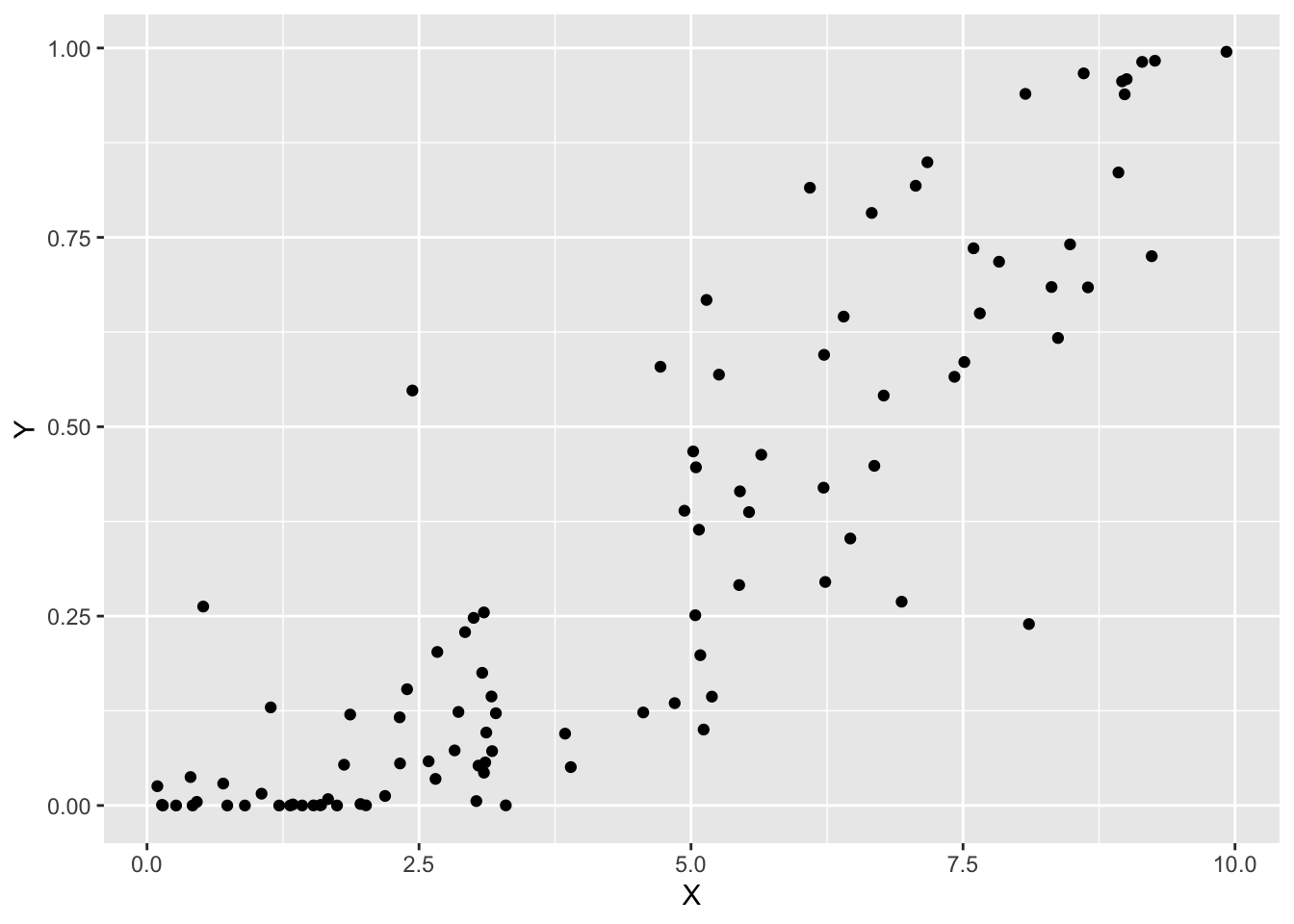
モデル
線形回帰
線形回帰を当てはめた場合です。
fit1 <- lm(Y ~ X, data = sim_data)
summary(fit1)
##
## Call:
## lm(formula = Y ~ X, data = sim_data)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.46227 -0.08929 -0.00822 0.08569 0.42536
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -0.126902 0.028096 -4.517 1.75e-05 ***
## X 0.102234 0.005424 18.848 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.1504 on 98 degrees of freedom
## Multiple R-squared: 0.7838, Adjusted R-squared: 0.7816
## F-statistic: 355.2 on 1 and 98 DF, p-value: < 2.2e-16結果を図示します。
p0 +
geom_abline(intercept = coef(fit1)[1],
slope = coef(fit1)[2])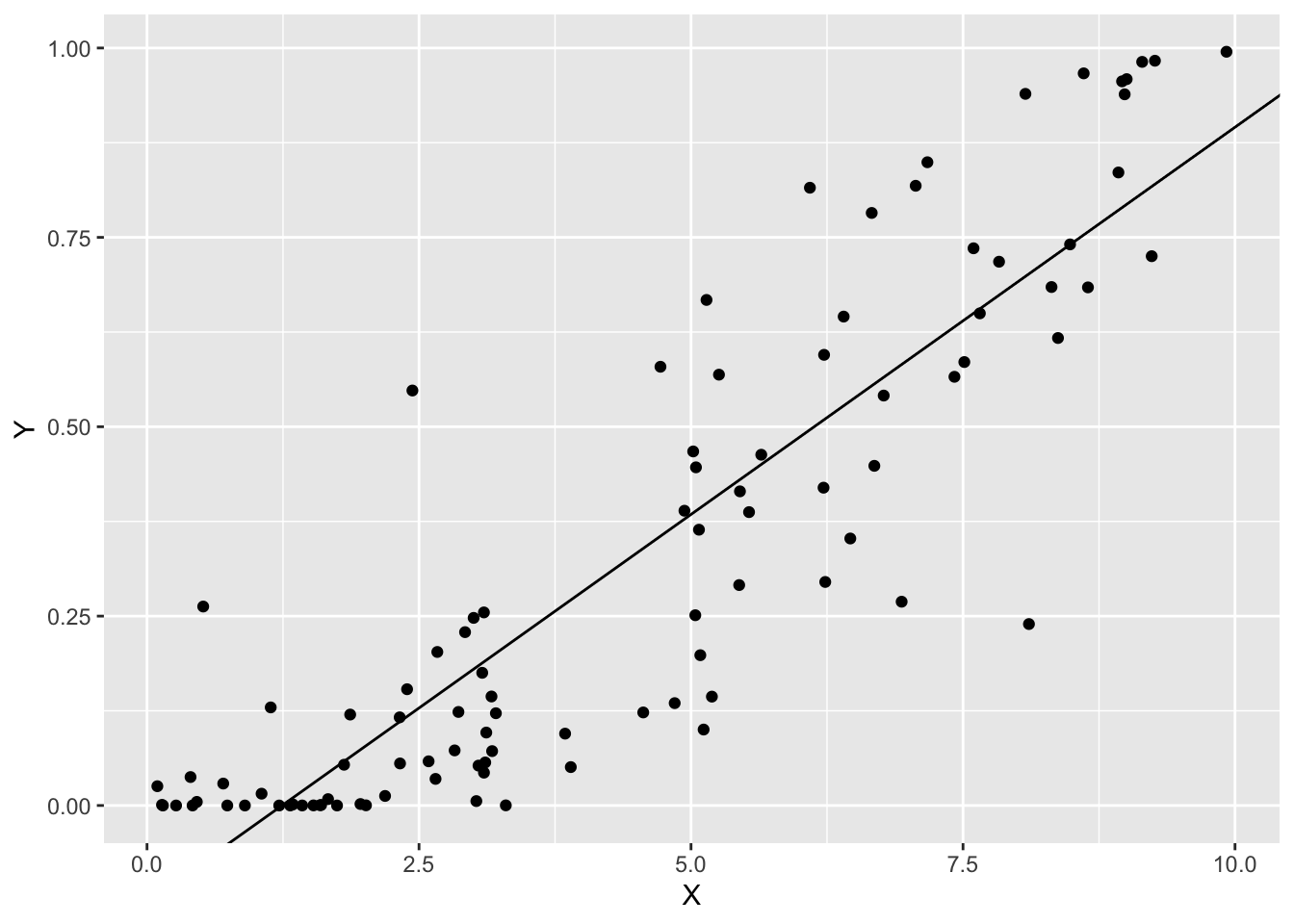
betaregパッケージを使ったベータ回帰
betaregパッケージを使った例です。
fit2 <- betareg(Y ~ X, sim_data, link = "logit")
summary(fit2)
##
## Call:
## betareg(formula = Y ~ X, data = sim_data, link = "logit")
##
## Quantile residuals:
## Min 1Q Median 3Q Max
## -2.7439 -0.6591 0.0894 0.8140 2.7383
##
## Coefficients (mean model with logit link):
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) -4.31787 0.24390 -17.70 <2e-16 ***
## X 0.68735 0.04227 16.26 <2e-16 ***
##
## Phi coefficients (precision model with identity link):
## Estimate Std. Error z value Pr(>|z|)
## (phi) 5.6091 0.8828 6.354 2.1e-10 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Type of estimator: ML (maximum likelihood)
## Log-likelihood: 259.4 on 3 Df
## Pseudo R-squared: 0.3637
## Number of iterations: 22 (BFGS) + 1 (Fisher scoring)結果を図示します。
p0 +
geom_function(fun = function(x)
inv_logit(coef(fit2)[1] + coef(fit2)[2] * x))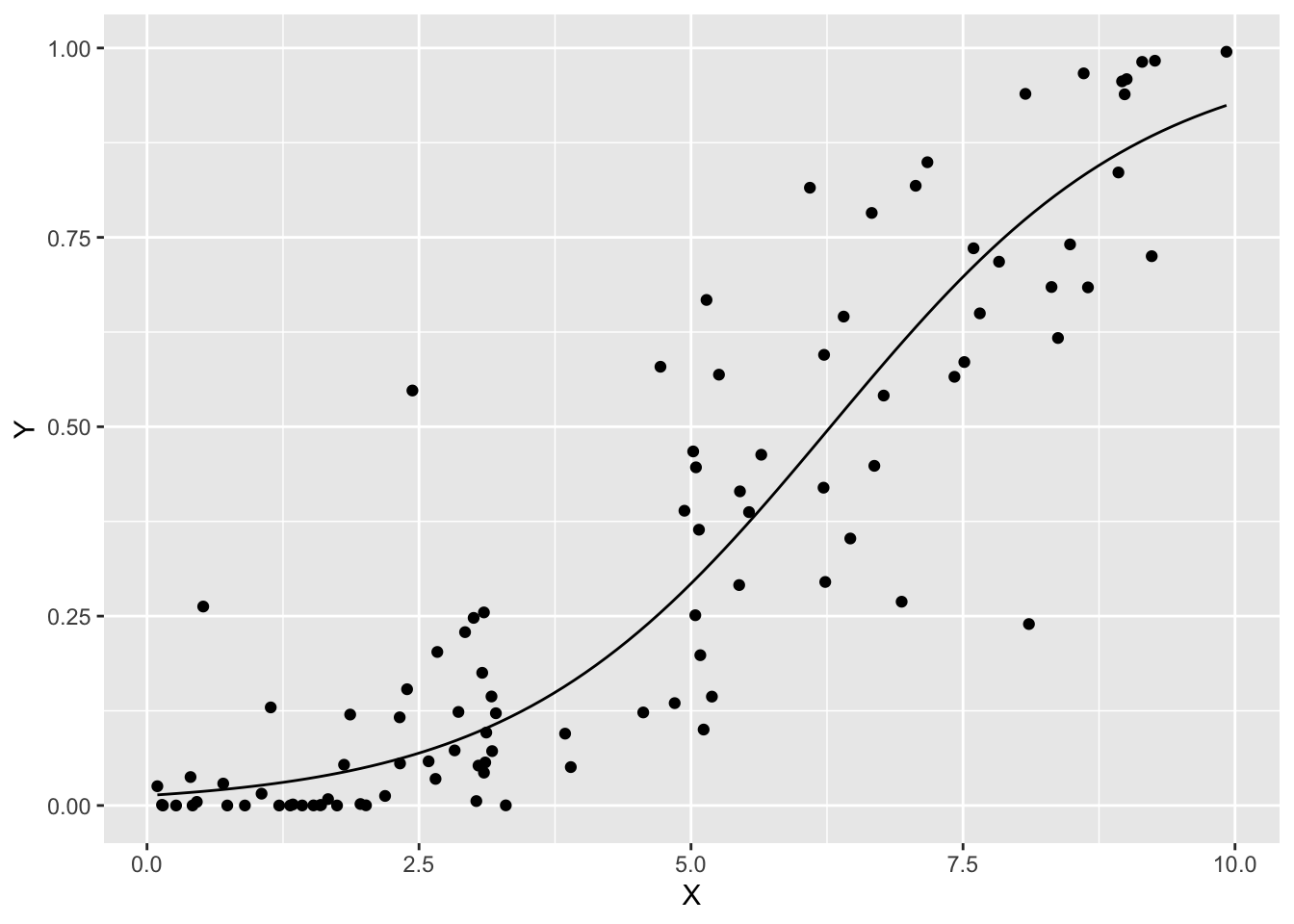
Stanを使ったベータ回帰
Stanを使った例です。
Stanのコードは以下のようになります。
data {
int<lower=0> N; // number of data points
vector[N] X; // explanatory variable
vector<lower=0,upper=1>[N] Y; // objective variable
}
parameters {
array[2] real beta; // intercept and slope (logit scale)
real<lower=0> kappa; // precision parameter
}
transformed parameters {
vector<lower=0,upper=1>[N] mu = inv_logit(beta[1] + beta[2] * X);
}
model {
Y ~ beta_proportion(mu, kappa);
// priors
beta ~ normal(0, 10);
}
generated quantities {
vector<lower=0,upper=1>[N] yrep;
for (n in 1:N)
yrep[n] = beta_proportion_rng(mu[n], kappa);
}上のStanコードをコンパイルしたものをmodelというオブジェクトに格納して、サンプリングをおこないます。
stan_data <- list(N = N, X = X, Y = Y)
fit3 <- model$sample(data = stan_data,
iter_warmup = 1000, iter_sampling = 1000,
refresh = 1000)
## Running MCMC with 4 sequential chains...
##
## Chain 1 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 1 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 1 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 1 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 1 finished in 0.2 seconds.
## Chain 2 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 2 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 2 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 2 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 2 finished in 0.2 seconds.
## Chain 3 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 3 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 3 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 3 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 3 finished in 0.2 seconds.
## Chain 4 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 4 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 4 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 4 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 4 finished in 0.2 seconds.
##
## All 4 chains finished successfully.
## Mean chain execution time: 0.2 seconds.
## Total execution time: 1.4 seconds.
fit3$summary(variables = c("beta", "kappa"))
## # A tibble: 3 × 10
## variable mean median sd mad q5 q95 rhat ess_bulk ess_tail
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 beta[1] -4.33 -4.33 0.224 0.227 -4.69 -3.95 1.00 1172. 1752.
## 2 beta[2] 0.689 0.690 0.0393 0.0389 0.623 0.753 1.00 1131. 1764.
## 3 kappa 5.67 5.63 0.841 0.857 4.34 7.09 1.00 1436. 1864.結果を図示します。
beta_mean <- fit3$summary("beta")$mean
p0 +
geom_function(fun = function(x)
inv_logit(beta_mean[1] + beta_mean[2] * x))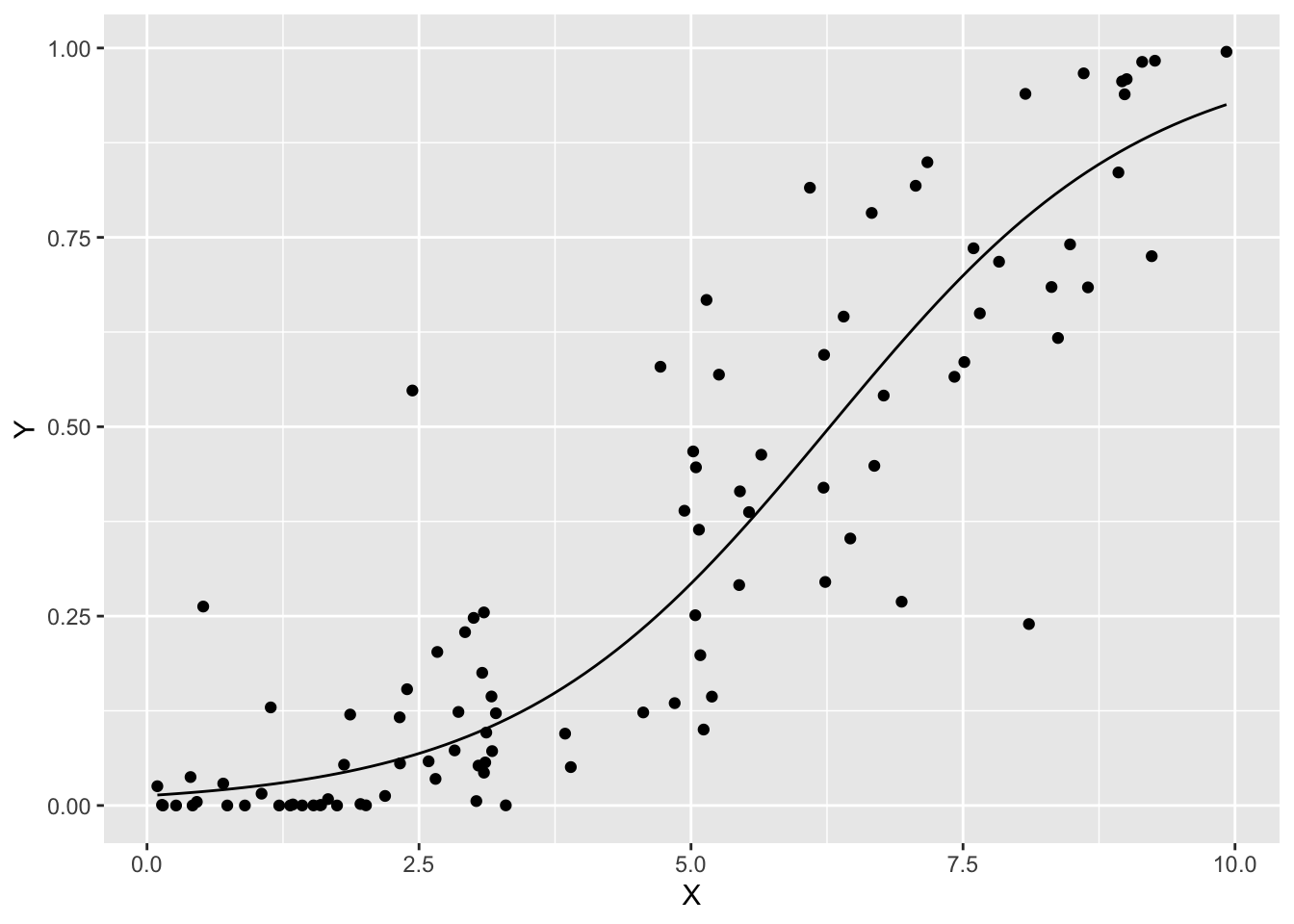
事後予測検査をしてみます。
yrep <- fit3$draws("yrep") |>
as_draws_matrix()
ppc_dens_overlay(y = Y, yrep = yrep[1:100, ])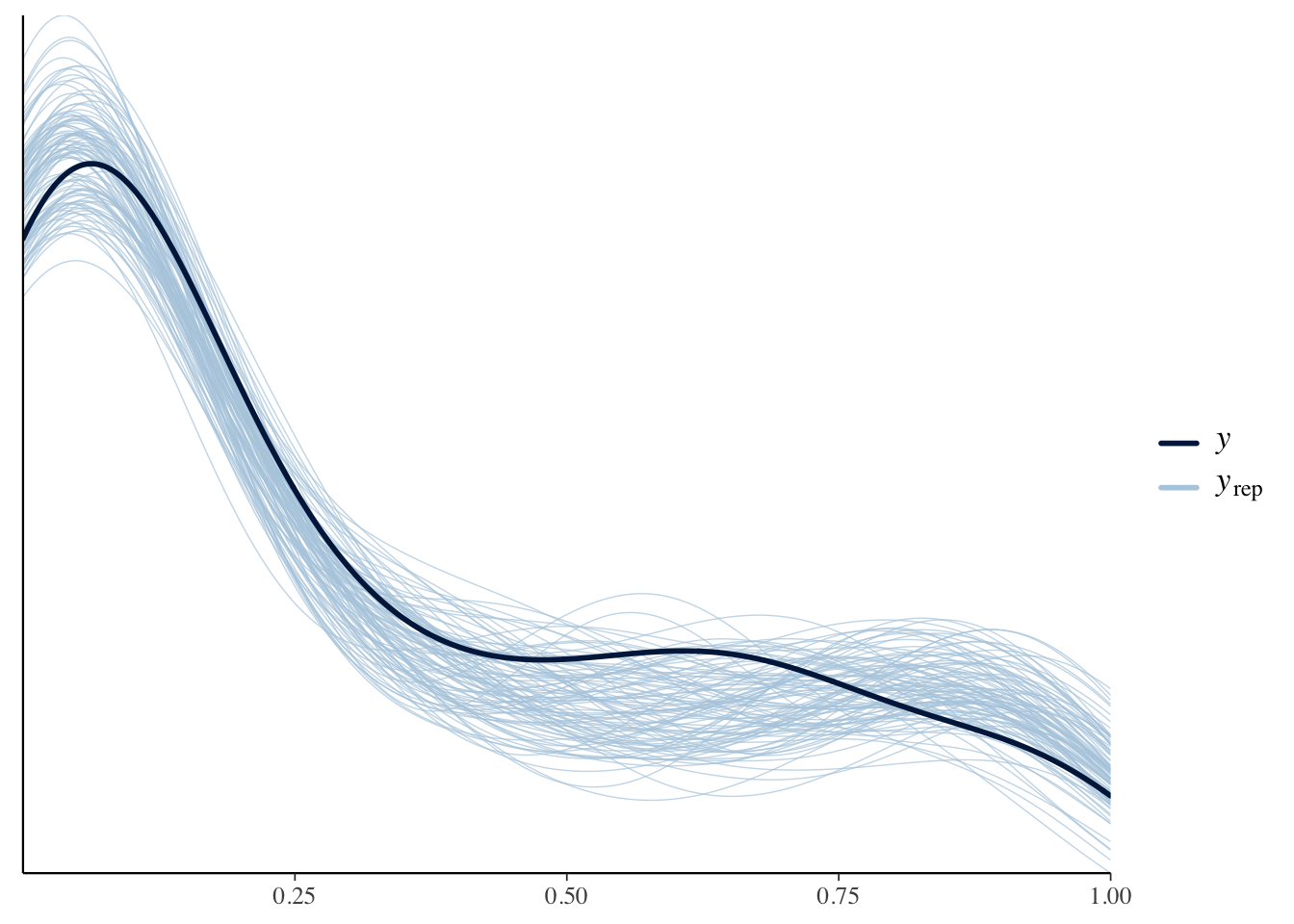
参考文献
- Imad Ali, Jonah Gabry and Ben Goodrich (2020) Modeling Rates/Proportions using Beta Regression with rstanarm. https://mc-stan.org/rstanarm/articles/betareg.html