library(readr)
library(dplyr)
##
## 次のパッケージを付け加えます: 'dplyr'
## 以下のオブジェクトは 'package:stats' からマスクされています:
##
## filter, lag
## 以下のオブジェクトは 'package:base' からマスクされています:
##
## intersect, setdiff, setequal, union
library(tidyr)
library(ggplot2)
data_url <- "https://figshare.com/ndownloader/files/40325779"森林の調査では、一定の範囲(方形区、ベルト、円形プロットなど)の中にある樹木の胸高直径1と樹高を測定するということがよくおこなわれます(毎木調査といいます)。こうした調査を長期間継続(毎年あるいは数年おきで)することにより、個々の樹木の成長や、枯死・新規加入の状況、森林全体の成長量などといったことがわかるようになります。
毎木調査のデータは一般に、個々の樹木を行に、測定項目(各年の胸高直径や樹高)を列に配置したスプレッドシートのデータとして保存されます2。新しいデータを測定したときには、その年の測定項目が列として追加され、新規加入の樹木が行に追加されます。いわゆる「横長」(wide)のデータです。これは整然(tidy)データではありません。
ということで、横長の毎木調査データを縦長(long)の整然データに変換するということをやってみます(最後には横長に戻すということもやってみます)。
データ
毎木調査データとして、石狩川源流地域における大規模風倒跡地の森林再生過程のデータのTable_S2.txtを使用します。データはfigshareにもあげてあり、以下のdata_urlに代入するURLからダウンロードできます。
データ形式
データの各列は以下のとおりです。各樹幹のデータが各行に はいっています。
- ID: 樹幹ID
- Individual_ID: 個体ID (同じ個体の樹幹は、同じ個体IDとなります)
- Belt: 個体の位置する調査ベルト番号
- X: 樹幹位置のベルト内のX座標 (m) (同じ個体でも、樹幹によって異なる場合があります)
- Y: 樹幹位置のベルト内のY座標 (m) (同じ個体でも、樹幹によって異なる場合があります)
- Scientific_name: 樹種の学名
- Japanese_name: 樹種の和名
- First: 樹幹が観測された最初の年
- Last: 樹幹が観測された最後の年 (生存幹は最後の調査年)
- H_1993: 1993年の樹高 (m); 欠測値はNA
- D_1993: 1993年の胸高直径 (cm)
- H_1998: 1998年の樹高 (m); 欠測値はNA
- D_1998: 1998年の胸高直径 (cm)
- H_2002: 2002年の樹高 (m)
- D_2002: 2002年の胸高直径 (cm)
- H_2009: 2009年の樹高 (m); 欠測値はNA
- D_2009: 2009年の胸高直径 (cm)
- H_2017: 2017年の樹高 (m)
- D_2017: 2017の胸高直径 (cm)
観測年より前あるいは枯死後の測定値もNAとしています。
データ読み込み
readrパッケージのread_tsv関数でデータを読み込みます。col_types引数でデータ型を指定しています。
data_orig <- read_tsv(data_url,
col_types = "ffiddcciidddddddddd")データの最初の6行をみてみます。
head(data_orig)
## # A tibble: 6 × 19
## ID Individual_ID Belt X Y Scientific_name Japanese_name First
## <fct> <fct> <int> <dbl> <dbl> <chr> <chr> <int>
## 1 1 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 2 2 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 3 3 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 4 4 4 27 5.9 0.3 Salix caprea バッコヤナギ 1993
## 5 5 4 27 5.9 0.3 Salix caprea バッコヤナギ 1993
## 6 6 4 27 5.9 0.3 Salix caprea バッコヤナギ 1993
## # ℹ 11 more variables: Last <int>, H_1993 <dbl>, D_1993 <dbl>, H_1998 <dbl>,
## # D_1998 <dbl>, H_2002 <dbl>, D_2002 <dbl>, H_2009 <dbl>, D_2009 <dbl>,
## # H_2017 <dbl>, D_2017 <dbl>wide→long変換
tidyrパッケージのpivot_longer関数で縦長(long)に変換します。
まずは、dplyrパッケージのselect関数で、IDとD_1993の2列のみを残したデータとしてから、どのように変換されるかをみてみます。dplyrパッケージのslice_head関数により、変換後の最初の10行を表示します。
data_orig %>%
select(ID, D_1993) %>%
pivot_longer(cols = "D_1993") %>%
slice_head(n = 10)
## # A tibble: 10 × 3
## ID name value
## <fct> <chr> <dbl>
## 1 1 D_1993 14.9
## 2 2 D_1993 8.3
## 3 3 D_1993 8.2
## 4 4 D_1993 12.5
## 5 5 D_1993 11.5
## 6 6 D_1993 11.2
## 7 7 D_1993 7.5
## 8 8 D_1993 10.3
## 9 9 D_1993 6.4
## 10 10 D_1993 7.6変換後のname列の内容がD_1993となっています。これをDと1993に分離して、前者をTypeという列に、後者をYearという列に格納するようにしてみます。
data_orig %>%
select(ID, D_1993) %>%
pivot_longer(cols = "D_1993",
names_to = c("Type", "Year"),
names_sep = "_",
values_to = "Value") %>%
slice_head(n = 10)
## # A tibble: 10 × 4
## ID Type Year Value
## <fct> <chr> <chr> <dbl>
## 1 1 D 1993 14.9
## 2 2 D 1993 8.3
## 3 3 D 1993 8.2
## 4 4 D 1993 12.5
## 5 5 D 1993 11.5
## 6 6 D 1993 11.2
## 7 7 D 1993 7.5
## 8 8 D 1993 10.3
## 9 9 D 1993 6.4
## 10 10 D 1993 7.6IDとすべての胸高直径データを残したデータについて、同様の処理をおこないます。dplyrパッケージのslice関数により、処理後のデータの最初の5行と最後の5行を表示するようにしてします。Year列に測定年が格納されているのが確認できます。
data_orig %>%
select(ID, starts_with("D_")) %>%
pivot_longer(cols = starts_with("D_"),
names_to = c("Type", "Year"),
names_sep = "_",
values_to = "Value") %>%
dplyr::slice((c(1:5, (n() - 4):n())))
## # A tibble: 10 × 4
## ID Type Year Value
## <fct> <chr> <chr> <dbl>
## 1 1 D 1993 14.9
## 2 1 D 1998 16.4
## 3 1 D 2002 17.9
## 4 1 D 2009 18.3
## 5 1 D 2017 19.1
## 6 574 D 1993 NA
## 7 574 D 1998 NA
## 8 574 D 2002 NA
## 9 574 D 2009 NA
## 10 574 D 2017 0.8上の結果には、観測年より前あるいは枯死後の測定値がNAとして含まれています。そこで、dplyrのfilter関数で、観測年より前あるいは枯死後の行を含めないようにします。それに先立ち、、dplyrのmutate関数で、Yearの型を文字列から整数に変換しています。
data_orig %>%
select(ID, First, Last, starts_with("D_")) %>%
pivot_longer(cols = starts_with("D_"),
names_to = c("Type", "Year"),
names_sep = "_",
values_to = "Value") %>%
mutate(Year = as.integer(Year)) %>%
filter(Year >= First & Year <= Last) %>%
slice((c(1:5, (n() - 4):n())))
## # A tibble: 10 × 6
## ID First Last Type Year Value
## <fct> <int> <int> <chr> <int> <dbl>
## 1 1 1993 2017 D 1993 14.9
## 2 1 1993 2017 D 1998 16.4
## 3 1 1993 2017 D 2002 17.9
## 4 1 1993 2017 D 2009 18.3
## 5 1 1993 2017 D 2017 19.1
## 6 570 2017 2017 D 2017 0.3
## 7 571 2017 2017 D 2017 1.1
## 8 572 2017 2017 D 2017 1.1
## 9 573 2017 2017 D 2017 0.9
## 10 574 2017 2017 D 2017 0.8さらに、樹高データも含めるようにします。結果のType列の値が’H’なのが、樹高データです。
data_orig %>%
select(ID, First, Last, starts_with("D_"), starts_with("H_")) %>%
pivot_longer(cols = starts_with("D_") | starts_with("H_"),
names_to = c("Type", "Year"),
names_sep = "_",
values_to = "Value") %>%
mutate(Year = as.integer(Year)) %>%
filter(Year >= First & Year <= Last) %>%
slice((c(1:5, (n() - 4):n())))
## # A tibble: 10 × 6
## ID First Last Type Year Value
## <fct> <int> <int> <chr> <int> <dbl>
## 1 1 1993 2017 D 1993 14.9
## 2 1 1993 2017 D 1998 16.4
## 3 1 1993 2017 D 2002 17.9
## 4 1 1993 2017 D 2009 18.3
## 5 1 1993 2017 D 2017 19.1
## 6 572 2017 2017 H 2017 1.4
## 7 573 2017 2017 D 2017 0.9
## 8 573 2017 2017 H 2017 1.4
## 9 574 2017 2017 D 2017 0.8
## 10 574 2017 2017 H 2017 1.4最後に、すべての列を含んだデータを縦長に変換し、data_longに格納するということをします。data_longの最初と最後の5行ずつを表示します。
data_long <- data_orig %>%
pivot_longer(cols = starts_with("D_") | starts_with("H_"),
names_to = c("Type", "Year"),
names_sep = "_",
values_to = "Value") %>%
mutate(Year = as.integer(Year)) %>%
filter(Year >= First & Year <= Last)
data_long %>%
slice((c(1:5, (n() - 4):n())))
## # A tibble: 10 × 12
## ID Individual_ID Belt X Y Scientific_name Japanese_name First
## <fct> <fct> <int> <dbl> <dbl> <chr> <chr> <int>
## 1 1 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 2 1 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 3 1 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 4 1 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 5 1 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 6 572 572 35 4.5 17.7 Abies sachalinensis トドマツ 2017
## 7 573 573 35 0.8 14.4 Picea glehnii アカエゾマツ 2017
## 8 573 573 35 0.8 14.4 Picea glehnii アカエゾマツ 2017
## 9 574 574 35 4.2 7.2 Picea glehnii アカエゾマツ 2017
## 10 574 574 35 4.2 7.2 Picea glehnii アカエゾマツ 2017
## # ℹ 4 more variables: Last <int>, Type <chr>, Year <int>, Value <dbl>ここまで、列名がD_1993やH_1998のようになっていて、区切り文字(ここでは”_“)が入っている場合を扱っていました。もし、D1993やH1998のように区切り文字のない場合は以下のように正規表現をつかって分離させることができます。
data_orig %>%
pivot_longer(cols = matches("[D|H][0-9]+"),
names_to = c("Type", "Year"),
names_pattern = "([D|H])([0-9]+)",
values_to = "Value")縦長データの利用
縦長の整然データは、ggplot2などでの扱いが容易になります。以下は、幹本数の多い3樹種について、平均胸高直径の時間変化をプロットするという例です。
spp <- data_long %>%
distinct(ID, Japanese_name) %>%
group_by(Japanese_name) %>%
tally() %>%
arrange(desc(n)) %>%
pull(Japanese_name)
data_long %>%
filter(Japanese_name %in% spp[1:3], Type == "D") %>%
group_by(Japanese_name, Year) %>%
summarise(Mean_dbh = mean(Value, na.rm = TRUE),
.groups = "drop") %>%
ggplot(aes(x = Year, y = Mean_dbh, colour = Japanese_name)) +
geom_line() +
geom_point(aes(shape = Japanese_name), size = 2) +
scale_x_continuous(name = "年",
limits = c(1990, 2020)) +
scale_y_continuous(name = "平均胸高直径 (cm)",
limits = c(5, 20)) +
scale_colour_discrete(name = "樹種") +
scale_shape_discrete(name = "樹種") +
theme_gray(base_family = "Source Han Sans")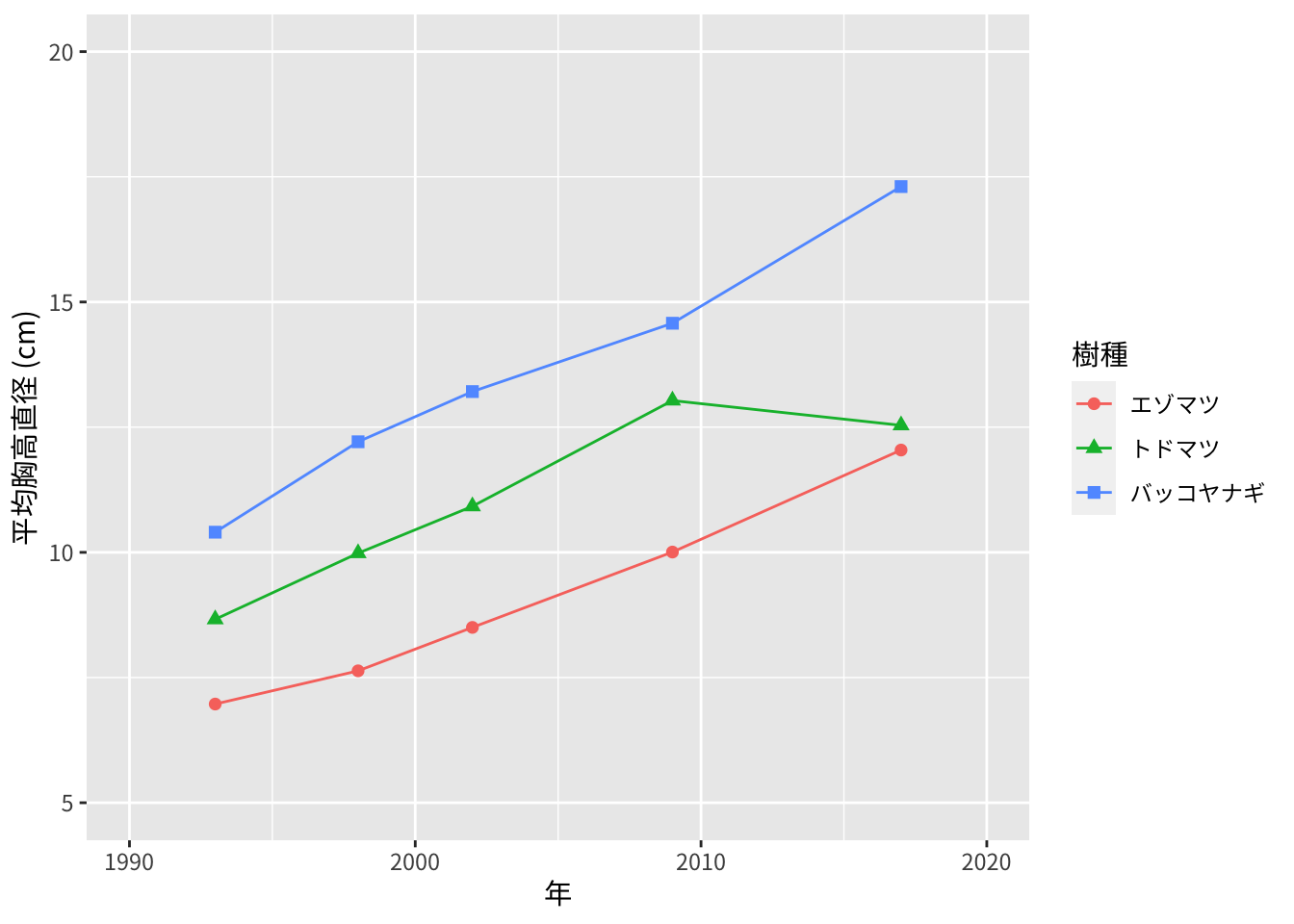
(フォントを”Source Han Sans”と指定していますが、適宜適当なフォントにしてください。)
long→wide変換
最後に、ふたたび横長(wide)データに戻すということをやってみます。これにはtidyrパッケージのpivot_wider関数を使用します。
まずは、ID, Type, Year, Valueの4列を残したデータで、変換するとどうなるかをしめします。変換後の横長データの最初と最後の5行ずつを表示します。
data_long %>%
select(ID, Type, Year, Value) %>%
pivot_wider(names_from = c(Type, Year), values_from = Value) %>%
slice((c(1:5, (n() - 4):n())))
## # A tibble: 10 × 11
## ID D_1993 D_1998 D_2002 D_2009 D_2017 H_1993 H_1998 H_2002 H_2009 H_2017
## <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 14.9 16.4 17.9 18.3 19.1 11 11.4 11.4 10.8 13.1
## 2 2 8.3 8.4 8.4 8.5 NA NA 7.3 6.7 6.5 NA
## 3 3 8.2 8.1 8.1 NA NA NA 6 5.6 NA NA
## 4 4 12.5 13.2 13.6 14.2 14.9 10.6 11.1 11.1 8.6 9.3
## 5 5 11.5 12.1 13 12.9 13.3 NA 11.6 10.8 8.8 8.1
## 6 570 NA NA NA NA 0.3 NA NA NA NA 1.4
## 7 571 NA NA NA NA 1.1 NA NA NA NA 2.1
## 8 572 NA NA NA NA 1.1 NA NA NA NA 1.4
## 9 573 NA NA NA NA 0.9 NA NA NA NA 1.4
## 10 574 NA NA NA NA 0.8 NA NA NA NA 1.4つづいて、データ丸ごとを横長に変換し、relocate関数で列の順番を並び替えたうえで、data_wideに格納します。前と同様に、data_wideの最初と最後の5行ずつを表示します(横に長すぎて省略されてしまいますが)。
data_wide <- data_long %>%
pivot_wider(names_from = c(Type, Year), values_from = Value) %>%
relocate(ends_with("1993"), ends_with("1998"), ends_with("2002"),
ends_with("2009"), ends_with("2017"), .after = Last)
data_wide %>%
slice((c(1:5, (n() - 4):n())))
## # A tibble: 10 × 19
## ID Individual_ID Belt X Y Scientific_name Japanese_name First
## <fct> <fct> <int> <dbl> <dbl> <chr> <chr> <int>
## 1 1 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 2 2 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 3 3 1 27 3.9 2.5 Betula ermanii ダケカンバ 1993
## 4 4 4 27 5.9 0.3 Salix caprea バッコヤナギ 1993
## 5 5 4 27 5.9 0.3 Salix caprea バッコヤナギ 1993
## 6 570 570 30 7 5.7 Abies sachalinensis トドマツ 2017
## 7 571 571 30 8.7 11.9 Abies sachalinensis トドマツ 2017
## 8 572 572 35 4.5 17.7 Abies sachalinensis トドマツ 2017
## 9 573 573 35 0.8 14.4 Picea glehnii アカエゾマツ 2017
## 10 574 574 35 4.2 7.2 Picea glehnii アカエゾマツ 2017
## # ℹ 11 more variables: Last <int>, D_1993 <dbl>, H_1993 <dbl>, D_1998 <dbl>,
## # H_1998 <dbl>, D_2002 <dbl>, H_2002 <dbl>, D_2009 <dbl>, H_2009 <dbl>,
## # D_2017 <dbl>, H_2017 <dbl>