library(palmerpenguins)
library(dplyr)
##
## 次のパッケージを付け加えます: 'dplyr'
## 以下のオブジェクトは 'package:stats' からマスクされています:
##
## filter, lag
## 以下のオブジェクトは 'package:base' からマスクされています:
##
## intersect, setdiff, setequal, union
library(ggplot2)1元配置分散分析を半分手動でやってみます。
データ
palmerpenguinsのデータを使って、bill_length_mmがspeciesにより異なるかどうかを検定します。
前処理
まず、missing completely at random (MCAR) を仮定して、欠測値のある行を除きます。
penguins2 <- penguins %>%
select(species, bill_length_mm) %>%
filter(!is.na(bill_length_mm))可視化
箱ヒゲ図にしてみます。
ggplot(penguins2) +
geom_boxplot(aes(x = species, y = bill_length_mm))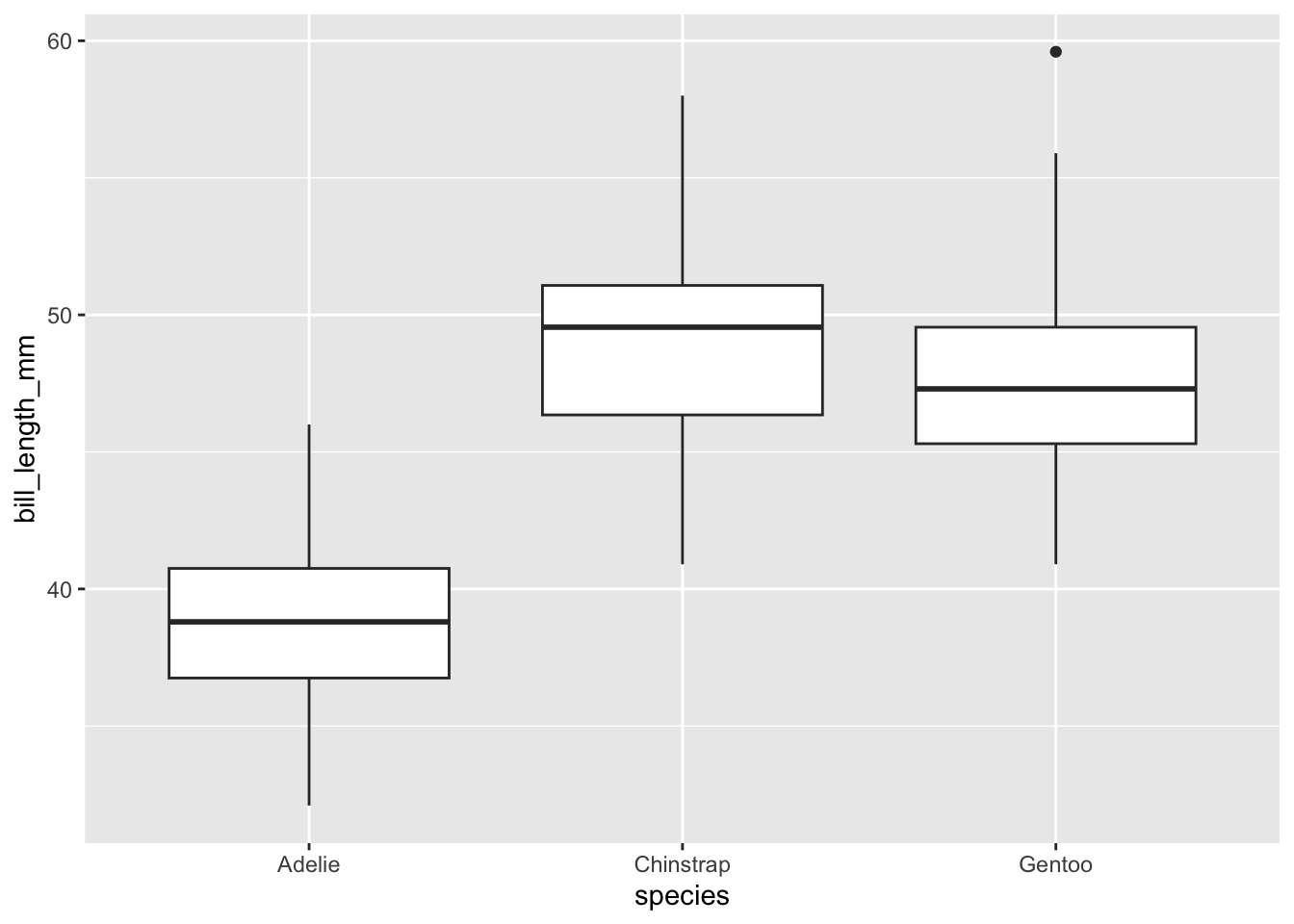
半分手動の分散分析
まず、全体平均を求めます。
(total_mean <- mean(penguins2$bill_length_mm))
## [1] 43.92193各種(水準)ごとの観測数(n)と平均(mean)、偏差平方和(sumsq)を求めます。
(penguins3 <- penguins2 %>%
group_by(species) %>%
summarise(n = n(),
mean = mean(bill_length_mm),
sumsq = n * (mean - total_mean)^2))
## # A tibble: 3 × 4
## species n mean sumsq
## <fct> <int> <dbl> <dbl>
## 1 Adelie 151 38.8 3975.
## 2 Chinstrap 68 48.8 1641.
## 3 Gentoo 123 47.5 1579.水準間平方和を求めます。
(sumsq_species <- penguins3 %>%
pull(sumsq) %>%
sum())
## [1] 7194.317残差平方和を求めます。
(sumsq_resid <- penguins2 %>%
left_join(penguins3, by = "species") %>%
mutate(sq_resid = (bill_length_mm - mean)^2) %>%
pull(sq_resid) %>%
sum())
## [1] 2969.888自由度を求めます。
(df_species <- levels(penguins2$species) %>%
length() - 1)
## [1] 2(df_resid <- nrow(penguins2) - df_species - 1)
## [1] 339平均平方を求めます。
(mean_sq_species <- sumsq_species / df_species)
## [1] 3597.159(mean_sq_resid <- sumsq_resid / df_resid)
## [1] 8.760732F値を求めます。
(F_value <- mean_sq_species / mean_sq_resid)
## [1] 410.6003最後にp値を求めます。
(pf(F_value, df_species, df_resid, lower.tail = FALSE))
## [1] 2.694614e-91aov関数による分散分析
aov関数の結果と比較してみます。
aov(bill_length_mm ~ species, data = penguins2) %>%
summary()
## Df Sum Sq Mean Sq F value Pr(>F)
## species 2 7194 3597 410.6 <2e-16 ***
## Residuals 339 2970 9
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1