library(cmdstanr)
## This is cmdstanr version 0.8.1
## - CmdStanR documentation and vignettes: mc-stan.org/cmdstanr
## - CmdStan path: /usr/local/cmdstan
## - CmdStan version: 2.35.0
library(bayesplot)
## This is bayesplot version 1.11.1
## - Online documentation and vignettes at mc-stan.org/bayesplot
## - bayesplot theme set to bayesplot::theme_default()
## * Does _not_ affect other ggplot2 plots
## * See ?bayesplot_theme_set for details on theme setting
library(ggplot2)
qgl <- read.csv(url("https://raw.githubusercontent.com/iwanami-datascience/vol1/master/ito/Qglauca.csv"))
register_knitr_engine()空間自己相関をとりいれたStanのモデルは、 Max Josephさんの“Exact sparse CAR models in Stan”、Mitzi Morrisさんの“Spatial Models in Stan: Intrinsic Auto-Regressive Models for Areal Data”がStanのCase Studiesに掲載されています。また松浦健太郎さんの“Bayesian Statistical Modeling with Stan, R, and Python”の第12章でも紹介されています。
今回はこのうちMitzi MorrisさんのICAR (Intrinsic Conditional Auto-Regressive) モデルを使ってみたいと思います。
データ
岩波データサイエンスVol.1で使用した、10×20のグリッド中のアラカシの株数データを使用します。
図示
まずはデータをプロットします。
ggplot(qgl, aes(x = X, y = Y)) +
geom_tile(aes(fill = N)) +
scale_y_reverse(breaks = c(0, 5, 10)) +
scale_fill_gradient(name = "株数", low = "grey90", high = "grey10") +
coord_fixed(ratio = 1) +
theme_bw(base_family = "IPAexGothic")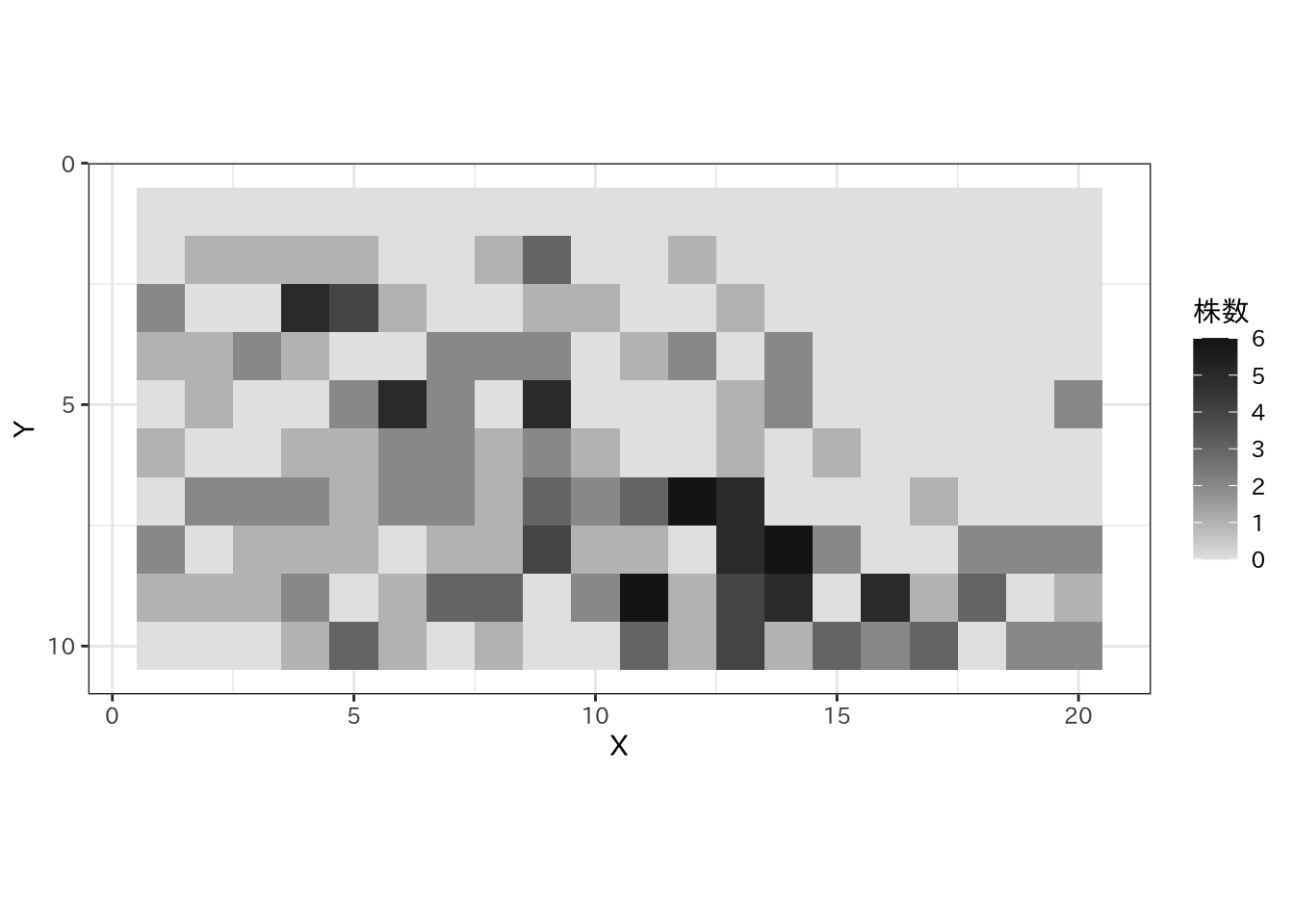
隣接リストの作成
隣接リストを作成します。このモデルでは、node1とnode2 (node1 < node2) に隣接関係を記述していきます。
n.x <- max(qgl$X)
n.y <- max(qgl$Y)
n_edges <- (2 * 4 +
3 * (2 * (n.x - 2) + (2 * (n.y - 2))) +
4 * (n.x - 2) * (n.y - 2)) / 2
node1 <- node2 <- rep(0, n_edges)
i <- 1
for (j in 1:(n.x - 1)) {
for (k in 1:(n.y - 1)) {
node1[i + 1] <- node1[i] <- n.y * (j - 1) + k
node2[i] <- n.y * (j - 1) + k + 1
node2[i + 1] <- n.y * j + k
i <- i + 2
}
}
# right side
for (j in 1:(n.y - 1)) {
node1[i] <- n.y * (n.x - 1) + j
node2[i] <- n.y * (n.x - 1) + j + 1
i <- i + 1
}
# bottom side
for (j in 1:(n.x - 1)) {
node1[i] <- n.y * j
node2[i] <- n.y * (j + 1)
i <- i + 1
}モデル
モデルです。説明変数は今回は用いないので、元のモデルにあったbeta1とxは消去してあります。その他はそのままです。
beta0が切片、phiが空間自己相関、thetaが不均一性のパラメータです。
/*
* from Spatial Models in Stan: Intrinsic Auto-Regressive Models for Areal Data
* https://mc-stan.org/users/documentation/case-studies/icar_stan.html
*/
data {
int<lower=0> N;
int<lower=0> N_edges;
array[N_edges] int<lower=1, upper=N> node1; // node1[i] adjacent to node2[i]
array[N_edges] int<lower=1, upper=N> node2; // and node1[i] < node2[i]
array[N] int<lower=0> Y; // count outcomes
}
parameters {
real beta0; // intercept
real<lower=0> tau_theta; // precision of heterogeneous effects
real<lower=0> tau_phi; // precision of spatial effects
vector[N] theta; // heterogeneous effects
vector[N] phi; // spatial effects
}
transformed parameters {
real<lower=0> sigma_theta = inv(sqrt(tau_theta)); // convert precision to sigma
real<lower=0> sigma_phi = inv(sqrt(tau_phi)); // convert precision to sigma
}
model {
Y ~ poisson_log(beta0 + phi * sigma_phi
+ theta * sigma_theta);
target += -0.5 * dot_self(phi[node1] - phi[node2]);
sum(phi) ~ normal(0, 0.001 * N); // equivalent to mean(phi) ~ normal(0,0.001)
beta0 ~ normal(0, 5);
theta ~ normal(0, 1);
tau_theta ~ gamma(3.2761, 1.81); // Carlin WinBUGS priors
tau_phi ~ gamma(1, 1); // Carlin WinBUGS priors
}
generated quantities {
vector[N] mu = exp(beta0 + phi * sigma_phi
+ theta * sigma_theta);
}あてはめ
cmdstanr経由でStanを実行してモデルをあてはめます。Stanのモデルはmodelオブジェクトにいれておいてあります。
stan_data <- list(N = nrow(qgl), N_edges = n_edges,
node1 = node1, node2 = node2,
Y = qgl$N)
fit <- model$sample(data = stan_data, seed = 123,
chains = 4, parallel_chains = 4,
iter_warmup = 1000, iter_sampling = 1000,
refresh = 1000)
## Running MCMC with 4 parallel chains...
##
## Chain 1 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 2 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 3 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 4 Iteration: 1 / 2000 [ 0%] (Warmup)
## Chain 2 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 2 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 4 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 4 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 1 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 1 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 3 Iteration: 1000 / 2000 [ 50%] (Warmup)
## Chain 3 Iteration: 1001 / 2000 [ 50%] (Sampling)
## Chain 2 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 2 finished in 5.0 seconds.
## Chain 4 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 4 finished in 6.2 seconds.
## Chain 1 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 1 finished in 6.4 seconds.
## Chain 3 Iteration: 2000 / 2000 [100%] (Sampling)
## Chain 3 finished in 6.7 seconds.
##
## All 4 chains finished successfully.
## Mean chain execution time: 6.1 seconds.
## Total execution time: 6.8 seconds.結果
beta0、tau_theta、tau_phiについて軌跡を確認します。各連鎖ともよく混ざっているようです。
as_draws(fit) |>
mcmc_trace(c("beta0", "tau_theta", "tau_phi"))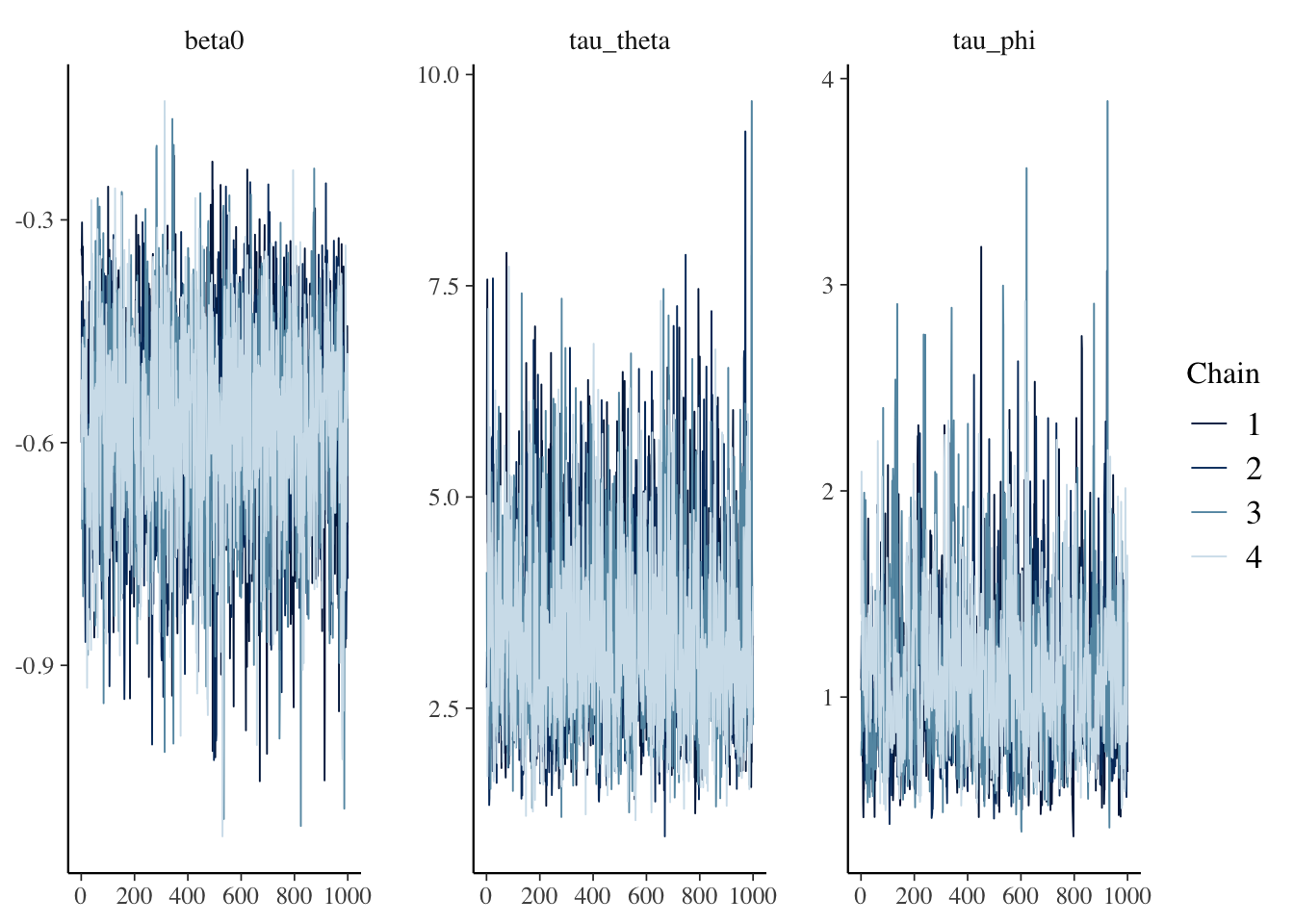
beta0とsigma_phi、sigma_thetaについて要約を表示します。
fit$summary(c("beta0", "sigma_phi", "sigma_theta"))
## # A tibble: 3 × 10
## variable mean median sd mad q5 q95 rhat ess_bulk ess_tail
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 beta0 -0.582 -0.575 0.132 0.127 -0.805 -0.376 1.00 2102. 2333.
## 2 sigma_phi 1.00 0.990 0.166 0.163 0.756 1.29 1.01 873. 1663.
## 3 sigma_theta 0.569 0.562 0.0902 0.0897 0.431 0.731 1.00 1817. 2385.phiについて要約を表示します。
fit$summary(c("phi"))
## # A tibble: 200 × 10
## variable mean median sd mad q5 q95 rhat ess_bulk ess_tail
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 phi[1] -0.668 -0.653 0.816 0.823 -2.01 0.665 1.00 2693. 2751.
## 2 phi[2] -0.378 -0.364 0.707 0.703 -1.55 0.767 1.00 2547. 2813.
## 3 phi[3] 0.179 0.203 0.628 0.627 -0.867 1.18 1.00 2213. 2938.
## 4 phi[4] 0.119 0.132 0.626 0.620 -0.924 1.12 1.00 2440. 2859.
## 5 phi[5] -0.111 -0.0910 0.642 0.632 -1.19 0.932 1.00 2848. 2982.
## 6 phi[6] 0.0157 0.0130 0.626 0.630 -1.01 1.06 1.00 2723. 2815.
## 7 phi[7] 0.0229 0.0152 0.629 0.630 -0.997 1.03 1.00 2721. 2751.
## 8 phi[8] 0.327 0.329 0.603 0.597 -0.698 1.30 1.00 2708. 3163.
## 9 phi[9] 0.0833 0.0983 0.644 0.652 -0.969 1.12 1.00 2257. 2931.
## 10 phi[10] -0.338 -0.322 0.786 0.790 -1.63 0.921 1.00 2562. 2859.
## # ℹ 190 more rowsthetaについて要約を表示します。
fit$summary(c("theta"))
## # A tibble: 200 × 10
## variable mean median sd mad q5 q95 rhat ess_bulk ess_tail
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 theta[1] -0.224 -0.223 0.946 0.949 -1.78 1.31 1.00 5023. 3076.
## 2 theta[2] -0.251 -0.230 0.928 0.929 -1.76 1.26 1.00 4424. 3193.
## 3 theta[3] 0.509 0.500 0.892 0.920 -0.955 1.97 1.00 3819. 3059.
## 4 theta[4] 0.0887 0.102 0.901 0.907 -1.40 1.57 1.00 4246. 3479.
## 5 theta[5] -0.332 -0.325 0.956 0.951 -1.91 1.26 0.999 5005. 3113.
## 6 theta[6] 0.133 0.135 0.934 0.946 -1.48 1.68 1.00 4527. 2554.
## 7 theta[7] -0.349 -0.354 0.917 0.909 -1.88 1.17 1.00 4828. 2882.
## 8 theta[8] 0.436 0.452 0.904 0.908 -1.08 1.90 1.00 4435. 3342.
## 9 theta[9] 0.0903 0.0892 0.939 0.915 -1.47 1.65 1.00 4367. 2786.
## 10 theta[10] -0.273 -0.276 0.963 0.980 -1.84 1.29 1.00 4478. 2759.
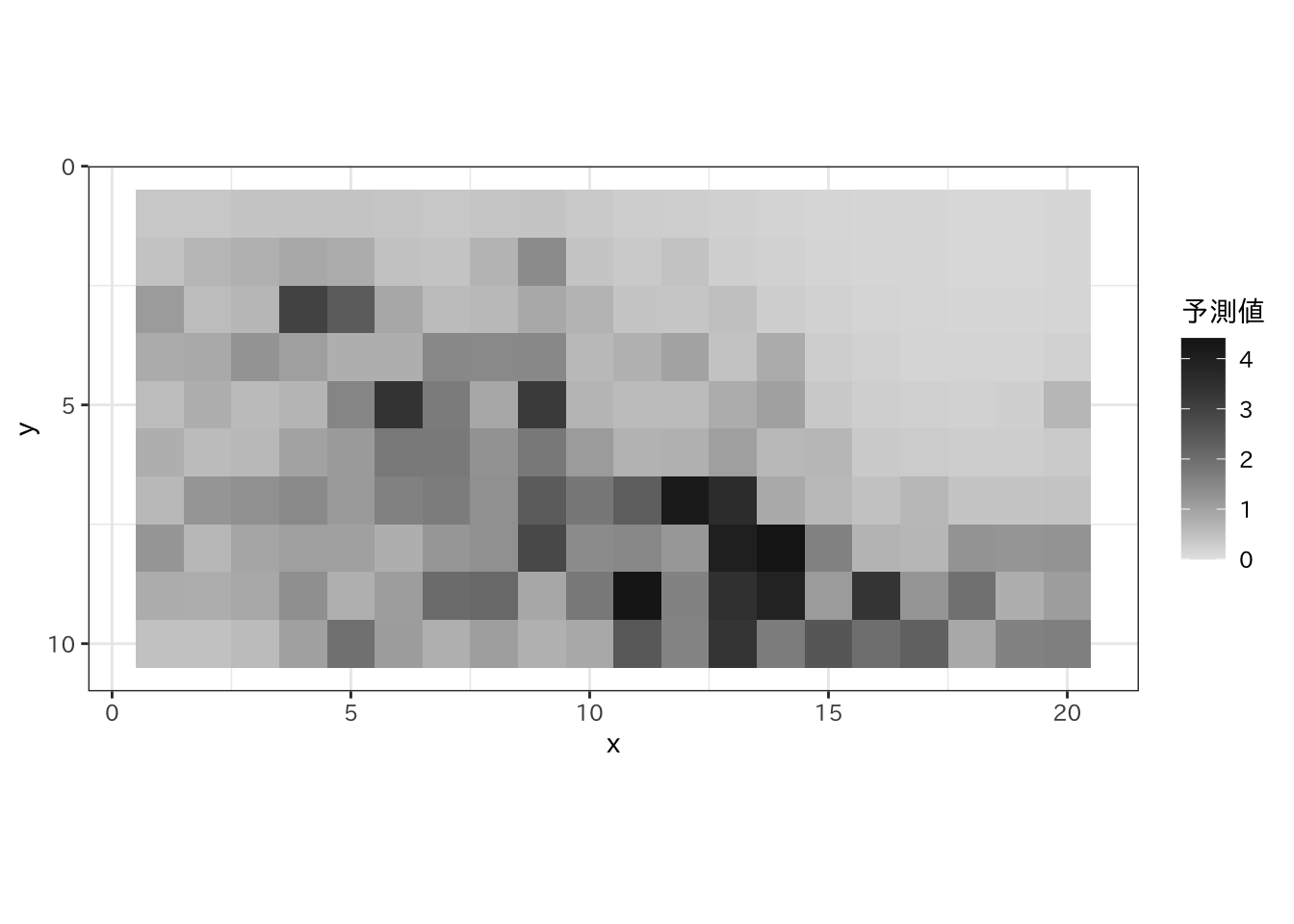
## # ℹ 190 more rows予測
各グリッドにおける株数の予測分布の平均値をプロットします。
fit$summary("mu") |>
dplyr::mutate(x = rep(1:n.x, each = n.y),
y = rep(1:n.y, n.x)) |>
ggplot(aes(x = x, y = y)) +
geom_tile(aes(fill = mean)) +
scale_y_reverse(breaks = c(0, 5, 10)) +
scale_fill_gradient(name = "予測値", limits = c(0, NA),
low = "grey90", high = "grey10") +
coord_fixed(ratio = 1) +
theme_bw(base_family = "IPAexGothic")