library(tidymodels)
## ── Attaching packages ────────────────────────────────────── tidymodels 1.3.0 ──
## ✔ broom 1.0.7 ✔ recipes 1.1.1
## ✔ dials 1.4.0 ✔ rsample 1.2.1
## ✔ dplyr 1.1.4 ✔ tibble 3.2.1
## ✔ ggplot2 3.5.1 ✔ tidyr 1.3.1
## ✔ infer 1.0.7 ✔ tune 1.3.0
## ✔ modeldata 1.4.0 ✔ workflows 1.2.0
## ✔ parsnip 1.3.0 ✔ workflowsets 1.1.0
## ✔ purrr 1.0.4 ✔ yardstick 1.3.2
## ── Conflicts ───────────────────────────────────────── tidymodels_conflicts() ──
## ✖ purrr::discard() masks scales::discard()
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ✖ recipes::step() masks stats::step()
library(palmerpenguins)
##
## 次のパッケージを付け加えます: 'palmerpenguins'
## 以下のオブジェクトは 'package:modeldata' からマスクされています:
##
## penguins
set.seed(123)Rで、tidymodelsを利用してサポートベクターマシンによる分類をためしてみました。tidymodelsの勉強には、著者のお一人の瓜生さんからいただいた下記書籍を参考にしました。
松村優哉・瓜生真也・吉村広志 Rユーザのためのtidymodels[実践]入門—モダンな統計・機械学習モデリングの世界 技術評論社
準備
tidymodelsパッケージを読み込みます。データにはpalmerpenguinsを使用しました。
データの加工
palmerpenguinsの3種のペンギンのうち、今回はアデリー (Adelie) とジェンツー (Gentoo) のみを使用して、bill_length_mmとbill_depth_mmの2つの測定値から両者の分類を試します。使用するデータだけを残してpenguins2というオブジェクトに代入しました。
penguins2 <- penguins |>
dplyr::filter(species %in% c("Adelie", "Gentoo")) |>
dplyr::mutate(species = factor(species,
levels = c("Adelie", "Gentoo"))) |>
dplyr::select(species, bill_length_mm, bill_depth_mm)データの確認
プロットしてデータを確認してみます。
penguins2 %>%
dplyr::filter(!is.na(bill_length_mm) &
!is.na(bill_depth_mm)) %>%
ggplot(aes(x = bill_length_mm, y = bill_depth_mm,
color = species)) +
geom_point()
レシピ
レシピの定義と前処理です。今回はデータ全体を使用することにして、学習用とテスト用とに分割するということはしていません。欠測値の補間はおこなっています。
penguins_recipe <- recipe(species ~ bill_length_mm + bill_depth_mm,
data = penguins2) |>
step_impute_mean(bill_length_mm, bill_depth_mm)モデル
線形サポートベクターマシーンを使用します。modeは分類として、engineにはkernlabを使用します。costには、デフォルトと同じ1を与えています。
svm_model <- svm_linear(mode = "classification",
engine = "kernlab",
cost = 1)ワークフロー
ワークフローのオブジェクトをつくって、レシピとモデルを追加します。
penguins_wf <- workflow() |>
add_recipe(penguins_recipe) |>
add_model(svm_model)あてはめ
あてはめを実行します。
penguins_fit <- penguins_wf |>
fit(data = penguins2)
## Setting default kernel parameters結果
結果を表示します。
penguins_fit |>
extract_fit_parsnip()
## parsnip model object
##
## Support Vector Machine object of class "ksvm"
##
## SV type: C-svc (classification)
## parameter : cost C = 1
##
## Linear (vanilla) kernel function.
##
## Number of Support Vectors : 10
##
## Objective Function Value : -6.17
## Training error : 0.007246
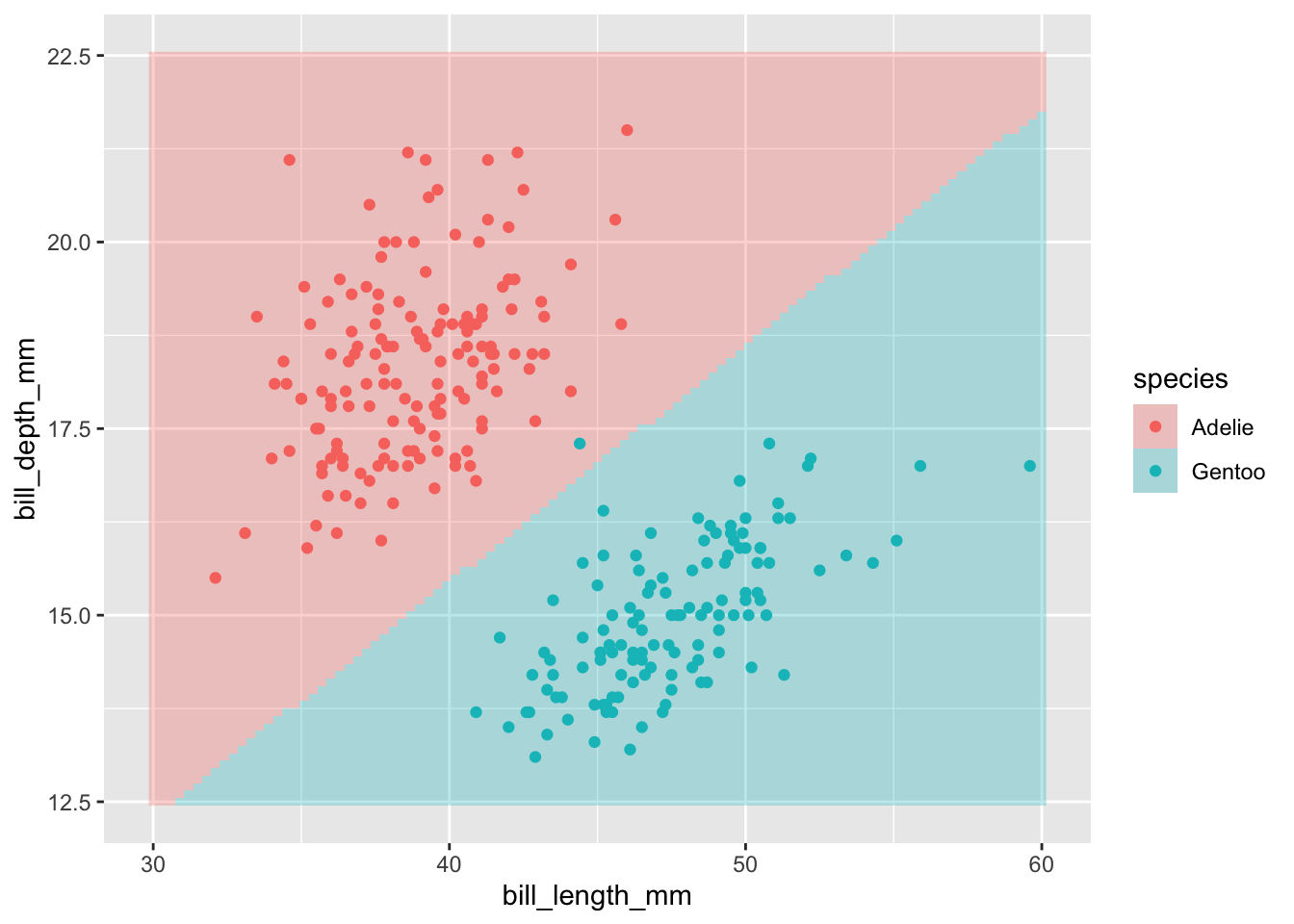
## Probability model included.判別境界の図示
グリッドで新データを生成して予測をおこない、判別境界を図示してみます。
new_data <- expand_grid(bill_length_mm = seq(30, 60, length = 101),
bill_depth_mm = seq(12.5, 22.5, length = 101))
penguins_predict <- augment(penguins_fit,
new_data = new_data) |>
dplyr::rename(species = .pred_class)
ggplot(penguins_predict,
aes(x = bill_length_mm, y = bill_depth_mm, fill = species)) +
geom_raster(alpha = 0.3) +
geom_point(data = dplyr::filter(penguins2,
!is.na(bill_length_mm) &
!is.na(bill_depth_mm)),
mapping = aes(x = bill_length_mm, y = bill_depth_mm,
colour = species))