library(tidymodels)
## ── Attaching packages ────────────────────────────────────── tidymodels 1.2.0 ──
## ✔ broom 1.0.5 ✔ recipes 1.0.10
## ✔ dials 1.2.1 ✔ rsample 1.2.1
## ✔ dplyr 1.1.4 ✔ tibble 3.2.1
## ✔ ggplot2 3.5.1 ✔ tidyr 1.3.1
## ✔ infer 1.0.7 ✔ tune 1.2.1
## ✔ modeldata 1.3.0 ✔ workflows 1.1.4
## ✔ parsnip 1.2.1 ✔ workflowsets 1.1.0
## ✔ purrr 1.0.2 ✔ yardstick 1.3.1
## ── Conflicts ───────────────────────────────────────── tidymodels_conflicts() ──
## ✖ purrr::discard() masks scales::discard()
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ✖ recipes::step() masks stats::step()
## • Use tidymodels_prefer() to resolve common conflicts.
library(discrim)
##
## 次のパッケージを付け加えます: 'discrim'
## 以下のオブジェクトは 'package:dials' からマスクされています:
##
## smoothness
library(palmerpenguins)
##
## 次のパッケージを付け加えます: 'palmerpenguins'
## 以下のオブジェクトは 'package:modeldata' からマスクされています:
##
## penguins
set.seed(123)前回の記事「tidymodelsからのサポートベクターマシン」のサポートベクターマシーンのところを判別分析に置き換えるということをやってみました。tidymodelsのおかげで、書き換えるところは最小限になっています。
準備
tidymodelsパッケージを読み込みます。今回は判別分析用にparsnipの補助パッケージであるdiscrimパッケージも読み込んでいます。
データにはやはりpalmerpenguinsを使用しています。
データの加工
使用するところだけを残して、新しいtibbleオブジェクトにします。前回と同じです。
penguins2 <- penguins |>
dplyr::filter(species %in% c("Adelie", "Gentoo")) |>
dplyr::mutate(species = factor(species,
levels = c("Adelie", "Gentoo"))) |>
dplyr::select(species, bill_length_mm, bill_depth_mm)データの確認
プロットしてデータを確認してみます。
penguins2 %>%
dplyr::filter(!is.na(bill_length_mm) &
!is.na(bill_depth_mm)) %>%
ggplot(aes(x = bill_length_mm, y = bill_depth_mm,
color = species)) +
geom_point()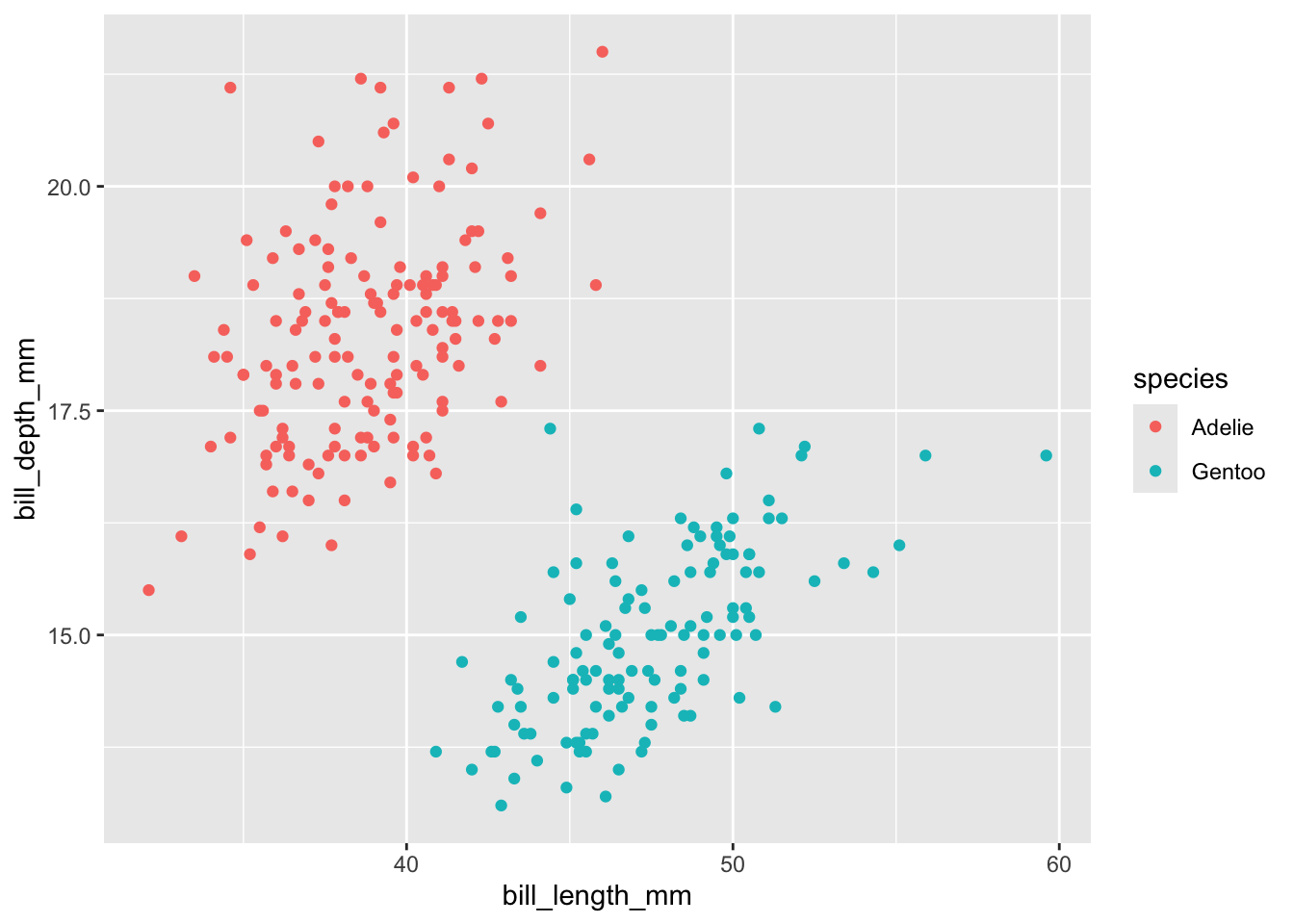
レシピ
ここも前回とまったく同じです。
recipe and preprocess
penguins_recipe <- recipe(species ~ bill_length_mm + bill_depth_mm,
data = penguins2) |>
step_impute_mean(bill_length_mm, bill_depth_mm)モデル1 (LDA)
まずは線形判別分析をモデルに使用します。MASSパッケージのldaが使われます。
lda_model <- discrim_linear(mode = "classification",
engine = "MASS")ワークフロー
add_modelの引数に、さきほど定義したlda_modelを割り当てます。
lda_wf <- workflow() |>
add_recipe(penguins_recipe) |>
add_model(lda_model)あてはめ
あてはめを実行します。
lda_fit <- lda_wf |>
fit(data = penguins2)結果
結果を表示します。
lda_fit |>
extract_fit_parsnip()
## parsnip model object
##
## Call:
## lda(..y ~ ., data = data)
##
## Prior probabilities of groups:
## Adelie Gentoo
## 0.5507246 0.4492754
##
## Group means:
## bill_length_mm bill_depth_mm
## Adelie 38.81712 18.33642
## Gentoo 47.46615 14.99707
##
## Coefficients of linear discriminants:
## LD1
## bill_length_mm 0.3380517
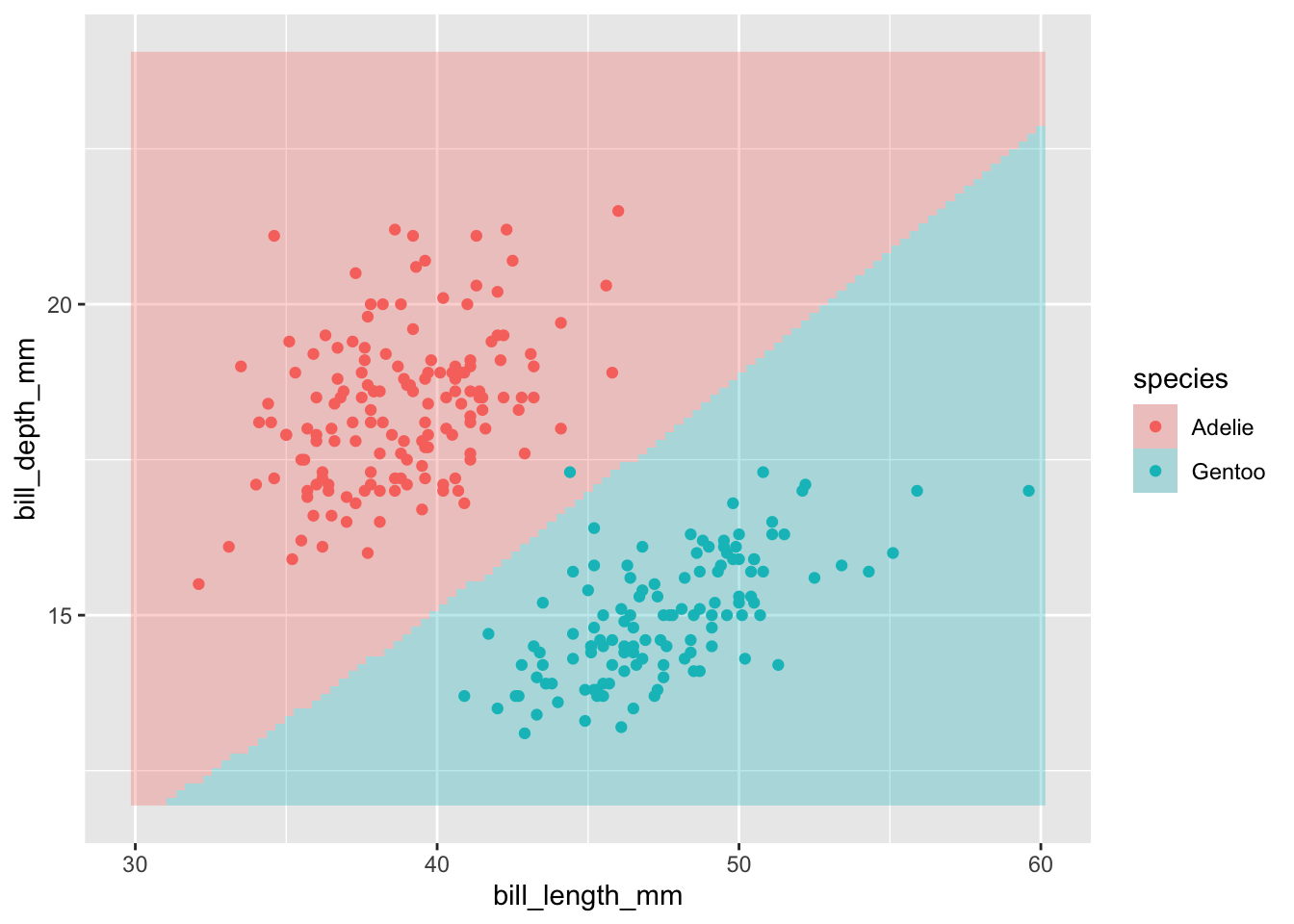
## bill_depth_mm -0.8614337判別境界の図示
グリッドで新データを生成して予測をおこない、判別境界を図示してみます。 ここも、オブジェクトがかわるだけで、コード自体はほぼ前回と同じです。
new_data <- expand_grid(bill_length_mm = seq(30, 60, length = 101),
bill_depth_mm = seq(12, 24, length = 101))
lda_predict <- augment(lda_fit,
new_data = new_data) |>
dplyr::rename(species = .pred_class)
ggplot(lda_predict,
aes(x = bill_length_mm, y = bill_depth_mm, fill = species)) +
geom_raster(alpha = 0.3) +
geom_point(data = dplyr::filter(penguins2,
!is.na(bill_length_mm) &
!is.na(bill_depth_mm)),
mapping = aes(x = bill_length_mm, y = bill_depth_mm,
colour = species))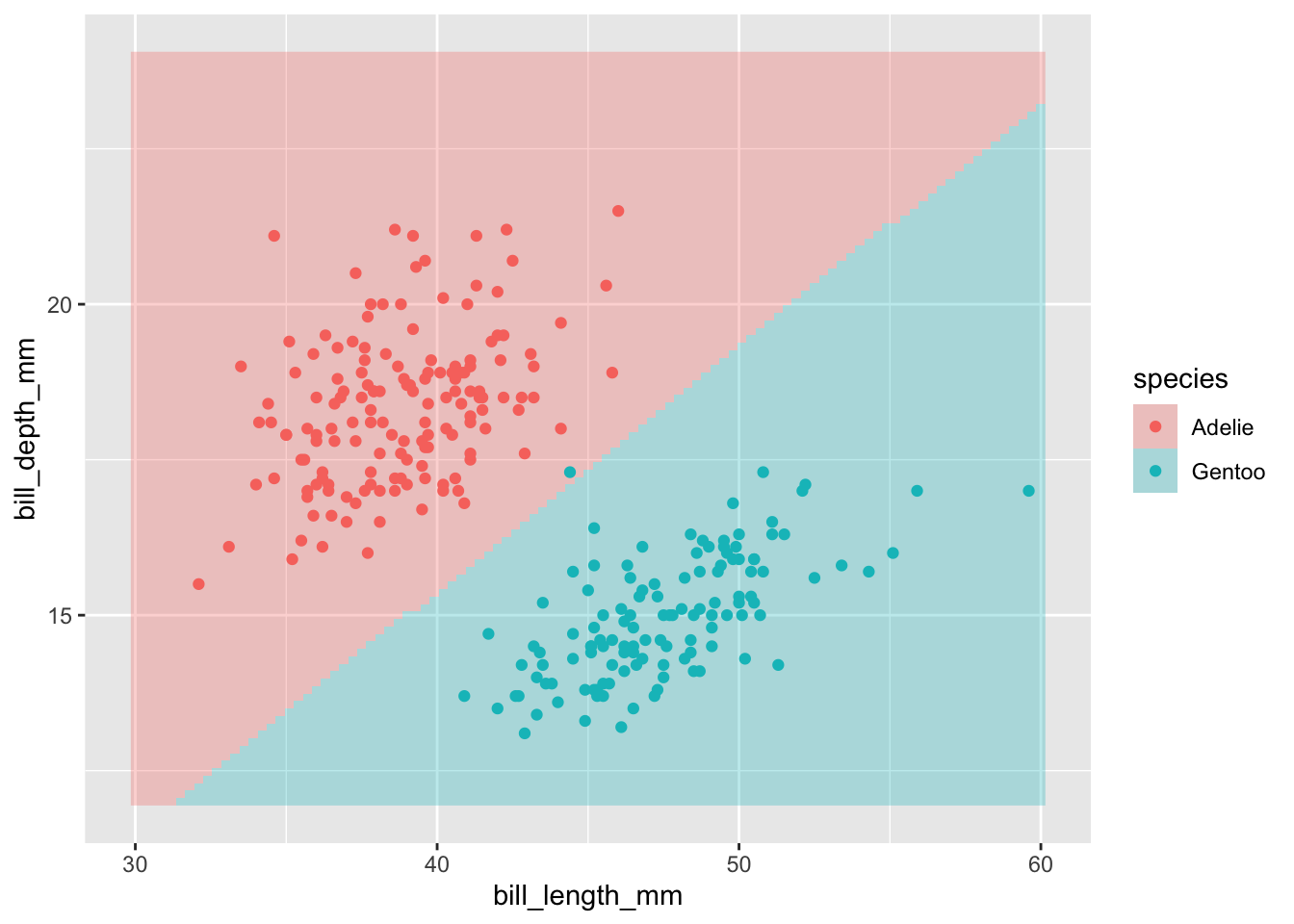
モデル2 (QDA)
次に二次判別分析を試します。MASSパッケージのqda関数が使われます。
qda_model <- discrim_quad(mode = "classification",
engine = "MASS")ワークフロー
ワークフローです。必要最小限の変更となっています。
qda_wf <- workflow() |>
add_recipe(penguins_recipe) |>
add_model(qda_model)あてはめ
あてはめを実行します。
qda_fit <- qda_wf |>
fit(data = penguins2)結果
結果です。
qda_fit |>
extract_fit_parsnip()
## parsnip model object
##
## Call:
## qda(..y ~ ., data = data)
##
## Prior probabilities of groups:
## Adelie Gentoo
## 0.5507246 0.4492754
##
## Group means:
## bill_length_mm bill_depth_mm
## Adelie 38.81712 18.33642
## Gentoo 47.46615 14.99707判別境界の図示
判別境界を図示してみます。よくみると、境界が曲線になっています。
new_data <- expand_grid(bill_length_mm = seq(30, 60, length = 101),
bill_depth_mm = seq(12, 24, length = 101))
qda_predict <- augment(qda_fit,
new_data = new_data) |>
dplyr::rename(species = .pred_class)
ggplot(qda_predict,
aes(x = bill_length_mm, y = bill_depth_mm, fill = species)) +
geom_raster(alpha = 0.3) +
geom_point(data = dplyr::filter(penguins2,
!is.na(bill_length_mm) &
!is.na(bill_depth_mm)),
mapping = aes(x = bill_length_mm, y = bill_depth_mm,
colour = species))