library(tidyverse)
## ── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
## ✔ dplyr 1.1.4 ✔ readr 2.1.5
## ✔ forcats 1.0.0 ✔ stringr 1.5.2
## ✔ ggplot2 4.0.0 ✔ tibble 3.3.0
## ✔ lubridate 1.9.4 ✔ tidyr 1.3.1
## ✔ purrr 1.1.0
## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors
library(vegan)
## Loading required package: permuteナラ枯れ1が北海道にまで本格的に上陸したようです。ナラ枯れなど撹乱があった広葉樹二次林で、その前後の樹種と本数がどのように変化したかを記録したデータがありますので、樹木の種多様度がどのように変化したのかを見ていこうと思います。
準備
パッケージを読み込みます。
データ
調査地
データは、Time series data of a broadleaved secondary forest in Japan as affected by deer and mass mortality of oak trees2 にて公開されているものです。
調査は、京都市左京区の銀閣寺山国有林に設定した1.05ha (210m×50m)の調査地で、1993年〜2014年の間(1993, 1996, 1999, 2002, 2005, 2014年)におこないました(1999年は欠測あり)。
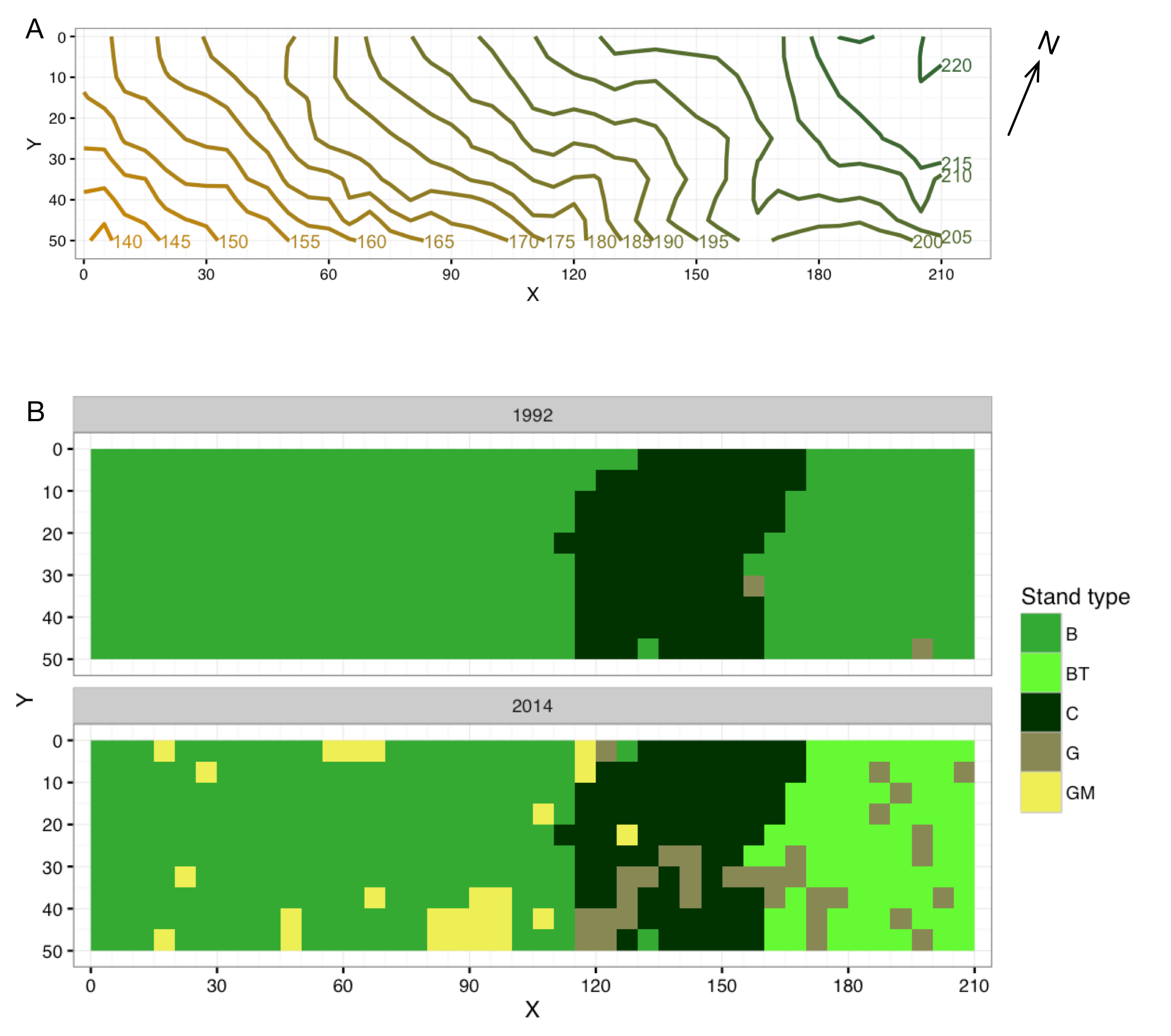
調査地の概要を 図 1 にしめします。Aは等高線を、Bは1993年と2014年の林相をしめしています。Bの林相の記号は以下のとおりです。
- B: 広葉樹林
- BT: 広葉樹林(間伐あり)
- C: 針葉樹林
- G: ギャップ
- GM: ナラ枯れによるギャップ
調査地を5m×5mの方形区に分割して、方形区ごとに樹木の樹種と本数を記録しました。
この調査地では、図の右側の針葉樹林と広葉樹林に間伐が入り、その後ナラ枯れが発生し、またシカの影響も増大するといった撹乱がはいりました。
そこで、1993年と2014年の測定結果間で、樹種の多様度がどのように変化したのかを検討します。
データ読み込み
公開されているデータ(Stem_data.csv)をダウンロードして、dataディレクトリに入れておきます。
read_csv関数で読み込みます。もとのデータの説明は論文にあるとおりです。方形区のIDとして、X座標とY座標の値を連結したものをQuadratという変数にして、最終的には、Quadrat、Species(樹種)、Start(観測開始年)、End(最後の生存年、2014年に生存していたものはNA)のみを残しています。
data_dir <- "data"
data_file <- "Stem_data.csv"
data <- read_csv(file.path(data_dir, data_file)) |>
dplyr::mutate(Quadrat = sprintf("%03d%02d", X, Y)) |>
dplyr::select(Quadrat, Species, Start, End)
## Rows: 4408 Columns: 9
## ── Column specification ────────────────────────────────────────────────────────
## Delimiter: ","
## chr (3): Indv, Stem, Species
## dbl (6): X, Y, X1, Y1, Start, End
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.データを確認します。
head(data, 10)
## # A tibble: 10 × 4
## Quadrat Species Start End
## <chr> <chr> <dbl> <dbl>
## 1 00000 Cleyera japonica 2014 NA
## 2 00000 Quercus glauca 2014 NA
## 3 00000 Symplocos prunifolia 1993 NA
## 4 00000 Gamblea innovans 1993 2005
## 5 00000 Gamblea innovans 1993 2002
## 6 00000 Quercus serrata 1993 NA
## 7 00000 Cleyera japonica 1993 1996
## 8 00005 Cleyera japonica 2014 NA
## 9 00005 Cleyera japonica 2014 NA
## 10 00005 Cleyera japonica 2014 NAデータ整形
解析用にデータを整形します。
あらかじめ、方形区ついて取りうる値の一覧をデータフレームとしてつくっておきます。
# Quadrat list
quad_df <- tidyr::expand_grid(X = seq(0, 205, 5),
Y = seq(0, 45, 5)) |>
dplyr:::mutate(Quadrat = sprintf("%03d%02d", X, Y)) |>
dplyr::select(Quadrat)1993年と2014のデータについて、方形区と種ごとに本数を合計したデータフレームを作成します。
data_1993 <- data |>
dplyr::filter(Start == 1993) |>
group_by(Quadrat, Species) |>
summarise(n = n(), .groups = "drop")
data_2014 <- data |>
dplyr::filter(is.na(End)) |>
group_by(Quadrat, Species) |>
summarise(n = n(), .groups = "drop")多様度の計算
種の豊富さ(種数)
まず、多様度の指標として種の豊富さ(種数)を計算します。これにはveganパッケージのspecnumber関数を使用しました。
方形区ごとに求めますが、1種も出現しなかった方形区もあるので、あらかじめ作成しておいた全方形区をふくむデータフレームに結合させる形で、データフレームdiversity_dfを作成します。
できたデータをさらにpivot_longerで変形してggplotのデータとして与え、ヒストグラムを作成します。
rich_1993 <- data_1993 |>
dplyr::group_by(Quadrat) |>
dplyr::summarise(nspc_1993 = specnumber(n))
rich_2014 <- data_2014 |>
dplyr::group_by(Quadrat) |>
dplyr::summarise(nspc_2014 = specnumber(n))
diversity_df <- quad_df |>
dplyr::left_join(rich_1993) |>
dplyr::left_join(rich_2014) |>
tidyr::replace_na(list(nspc_1993 = 0,
nspc_2014 = 0))
## Joining with `by = join_by(Quadrat)`
## Joining with `by = join_by(Quadrat)`
diversity_df |>
tidyr::pivot_longer(cols = nspc_1993:nspc_2014,
names_sep = "_",
names_to = c("Metric", "Year")) |>
ggplot(aes(x = value, fill = Year, group = Year)) +
geom_bar(position = "dodge") +
labs(x = "種数", y = "方形区数") +
scale_fill_discrete(name = "年") +
theme_gray(base_family = "Noto Sans JP")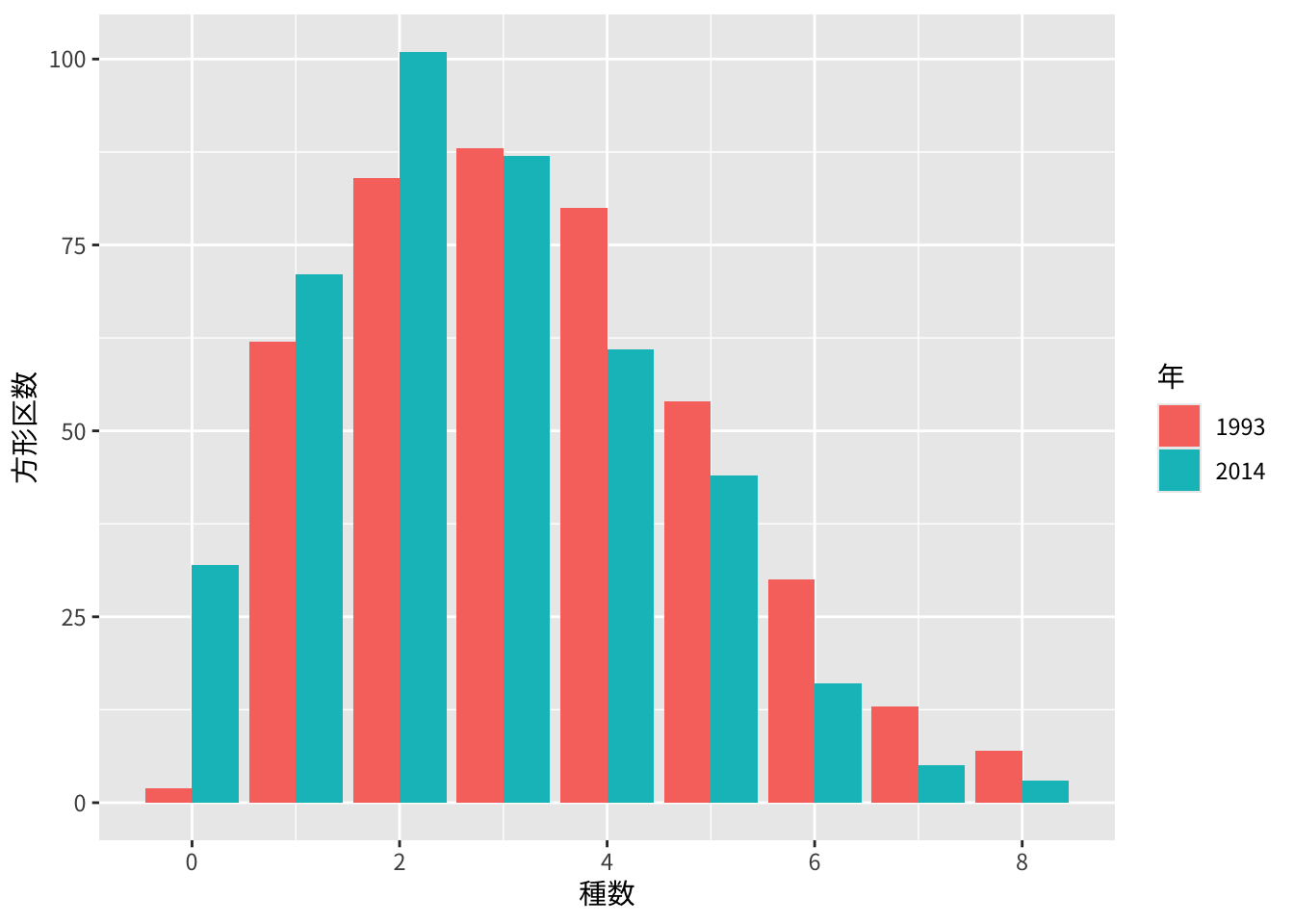
要約をしめします。
summary(diversity_df)
## Quadrat nspc_1993 nspc_2014
## Length:420 Min. :0.00 Min. :0.000
## Class :character 1st Qu.:2.00 1st Qu.:2.000
## Mode :character Median :3.00 Median :3.000
## Mean :3.36 Mean :2.745
## 3rd Qu.:4.00 3rd Qu.:4.000
## Max. :8.00 Max. :8.000四分位数は同じですが、平均が2014年では小さくなったようです。
対応のあるWilcoxon検定で比較してみます。
wilcox.test(diversity_df$nspc_1993, diversity_df$nspc_2014,
paired = TRUE)
##
## Wilcoxon signed rank test with continuity correction
##
## data: diversity_df$nspc_1993 and diversity_df$nspc_2014
## V = 35116, p-value = 1.989e-11
## alternative hypothesis: true location shift is not equal to 0有意な差がありました。2014年のほうが方形区単位では種数が少なくなったようです。
Shannonの多様度指数
つづいて、Shannonの多様度指数を求めます。これにはveganパッケージのdiversity関数を使用します。まずはこちらも方形区ごとにもとめました。
div_1993 <- data_1993 |>
dplyr::group_by(Quadrat) |>
dplyr::summarise(shannon_1993 = diversity(n, index = "shannon"))
div_2014 <- data_2014 |>
dplyr::group_by(Quadrat) |>
dplyr::summarise(shannon_2014 = diversity(n, index = "shannon"))
diversity_df <- diversity_df |>
dplyr::left_join(div_1993) |>
dplyr::left_join(div_2014)
## Joining with `by = join_by(Quadrat)`
## Joining with `by = join_by(Quadrat)`Shannon指数も加えたdiversity_dfデータフレームの要約です。Shannon指数も2014年には小さくなったようです。なお、1種も出現しなかったところはShannon指数はNAになっています。
summary(diversity_df)
## Quadrat nspc_1993 nspc_2014 shannon_1993
## Length:420 Min. :0.00 Min. :0.000 Min. :0.0000
## Class :character 1st Qu.:2.00 1st Qu.:2.000 1st Qu.:0.6365
## Mode :character Median :3.00 Median :3.000 Median :1.0397
## Mean :3.36 Mean :2.745 Mean :0.9550
## 3rd Qu.:4.00 3rd Qu.:4.000 3rd Qu.:1.3863
## Max. :8.00 Max. :8.000 Max. :2.0253
## NA's :2
## shannon_2014
## Min. :0.0000
## 1st Qu.:0.5623
## Median :0.9267
## Mean :0.8317
## 3rd Qu.:1.2411
## Max. :1.9513
## NA's :32図示します。
diversity_df |>
tidyr::pivot_longer(cols = nspc_1993:shannon_2014,
names_sep = "_",
names_to = c("Metric", "Year")) |>
dplyr::filter(Metric == "shannon",
!is.na(value)) |>
ggplot(aes(x = value, group = Year, fill = Year)) +
geom_histogram(binwidth = 0.25, position = "dodge") +
labs(x = "種数", y = "方形区数") +
scale_fill_discrete(name = "年") +
theme_gray(base_family = "Noto Sans JP")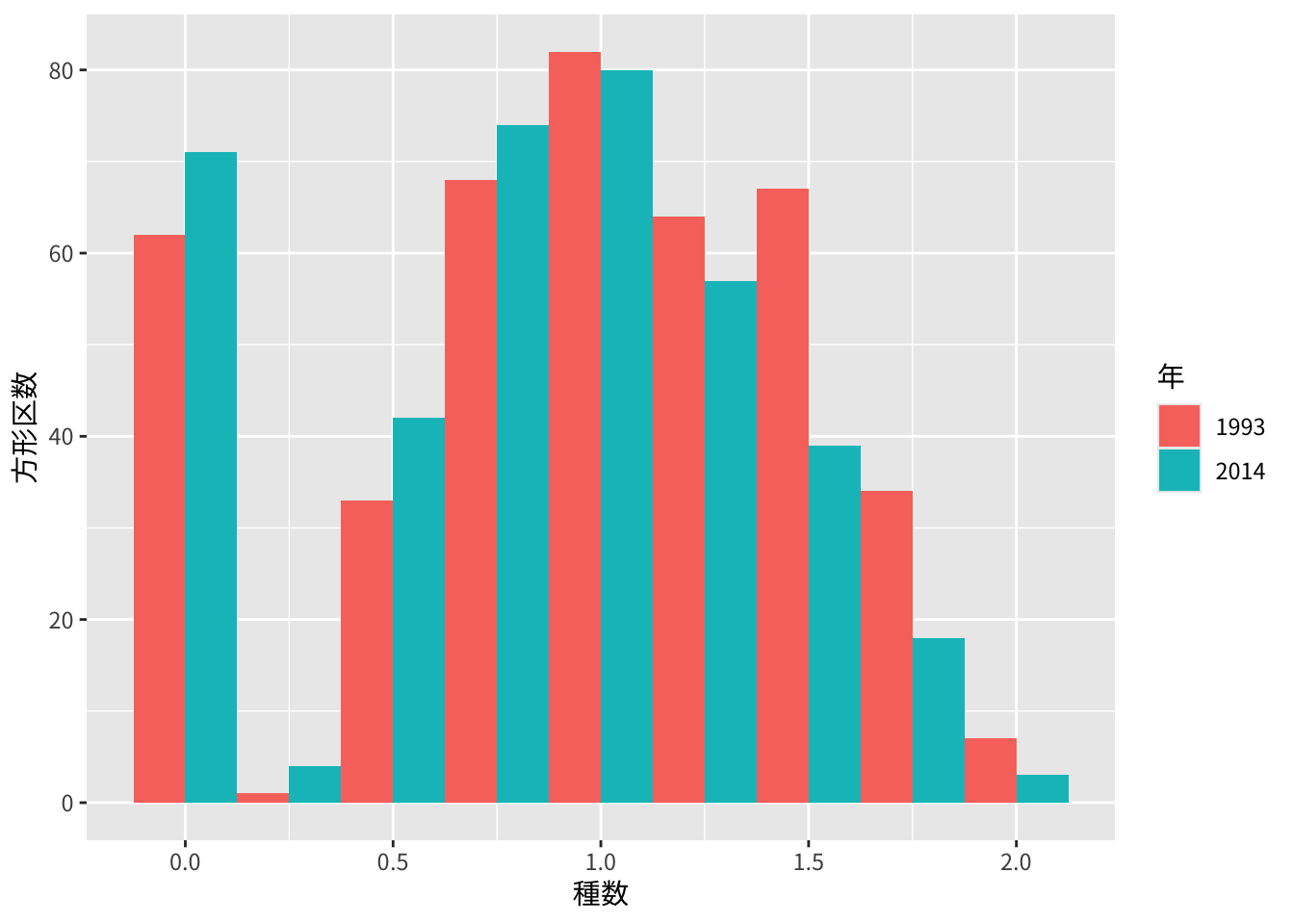
対応のあるWilcoxon検定で比較してみます。
wilcox.test(diversity_df$shannon_1993, diversity_df$shannon_2014,
paired = TRUE)
##
## Wilcoxon signed rank test with continuity correction
##
## data: diversity_df$shannon_1993 and diversity_df$shannon_2014
## V = 39868, p-value = 6.901e-06
## alternative hypothesis: true location shift is not equal to 0有意な差がありました。多様度は小さくなったようです。
調査地全体の多様度
最後に、調査地をまとめてShannonの多様度指数を求めます。
data_1993 |>
dplyr::group_by(Species) |>
dplyr::summarise(n = n(), .groups = "drop") |>
dplyr::summarise(shannon = diversity(n, index = "shannon"))
## # A tibble: 1 × 1
## shannon
## <dbl>
## 1 3.13
data_2014 |>
dplyr::group_by(Species) |>
dplyr::summarise(n = n(), .groups = "drop") |>
dplyr::summarise(shannon = diversity(n, index = "shannon"))
## # A tibble: 1 × 1
## shannon
## <dbl>
## 1 2.85こちらも、2014年は小さくなったようです。
おわりに
2014年のほうが1993年よりも多様度が小さくなったようです。
原因を探るためには、どこで小さくなったかが問題になるでしょう。ということで、Shannonの多様度指数の差をプロットしてみました。
diversity_df |>
dplyr::mutate(X = str_sub(Quadrat, 1, 3) |> as.numeric(),
Y = str_sub(Quadrat, 4, 5) |> as.numeric(),
diff = shannon_2014 - shannon_1993) |>
ggplot(aes(x = X, y = Y, fill = diff)) +
geom_tile() +
coord_fixed() +
scale_fill_continuous(name = "多様度の差", palette = "viridis") +
scale_y_reverse() +
theme_bw(base_family = "Noto Sans JP") +
theme(legend.position = "bottom")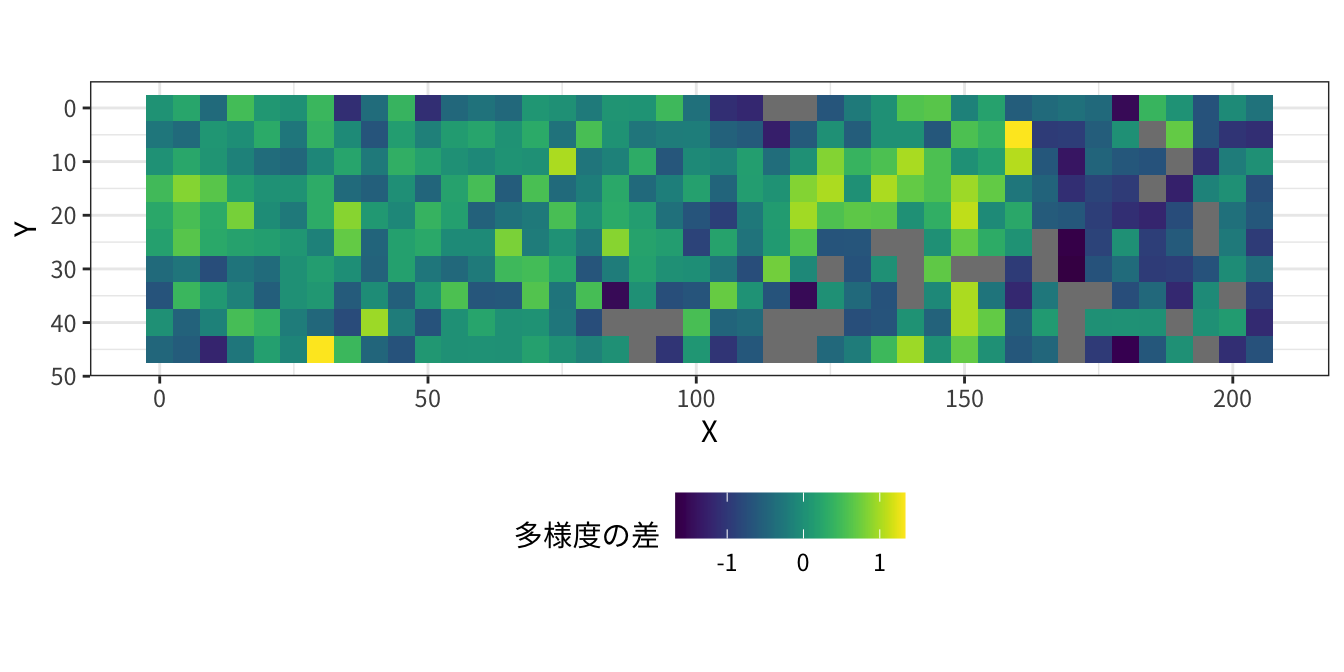
これを見ると、間伐を受けた広葉樹林のところで多様度が減少しているように思われます。ナラ枯れのほうはそれよりも影響は大きくなさそうです。