data(cancer, package = "survival")
head(aml)
## time status x
## 1 9 1 Maintained
## 2 13 1 Maintained
## 3 13 0 Maintained
## 4 18 1 Maintained
## 5 23 1 Maintained
## 6 28 0 Maintainedカプラン=マイヤー曲線をggplot2を使用して描く方法としては、survminerパッケージのggsurvplot関数を使う方法や、jskmパッケージのjskm関数を使う方法もありますが、今回はggplot2.utilsパッケージのgeom_km関数を使う方法をやってみました。
データ
survivalパッケージのamlを使用します。
変数は以下のとおりです。
time: 生存または打ちきり時間(単位は週らしい)status: 打ちきりの状態- 0は生存(打ちきり)、1が死亡
x: 維持化学療法の有無- Maintained: あり、Nonmaintained: なし
描画
geom_km関数で、カプラン=マイヤー曲線を描画します。mappingに与えるaes関数では、timeに生存時間を、statusに状態(0=生存, 1=死亡)を指定します。また、color = xとして、維持化学療法の有無の有無により色を分けた曲線とするようにします。
library(ggplot2.utils)
## Loading required package: ggplot2
## Registered S3 methods overwritten by 'ggpp':
## method from
## heightDetails.titleGrob ggplot2
## widthDetails.titleGrob ggplot2
ggplot(data = aml,
mapping = aes(time = time, status = status, color = x)) +
geom_km()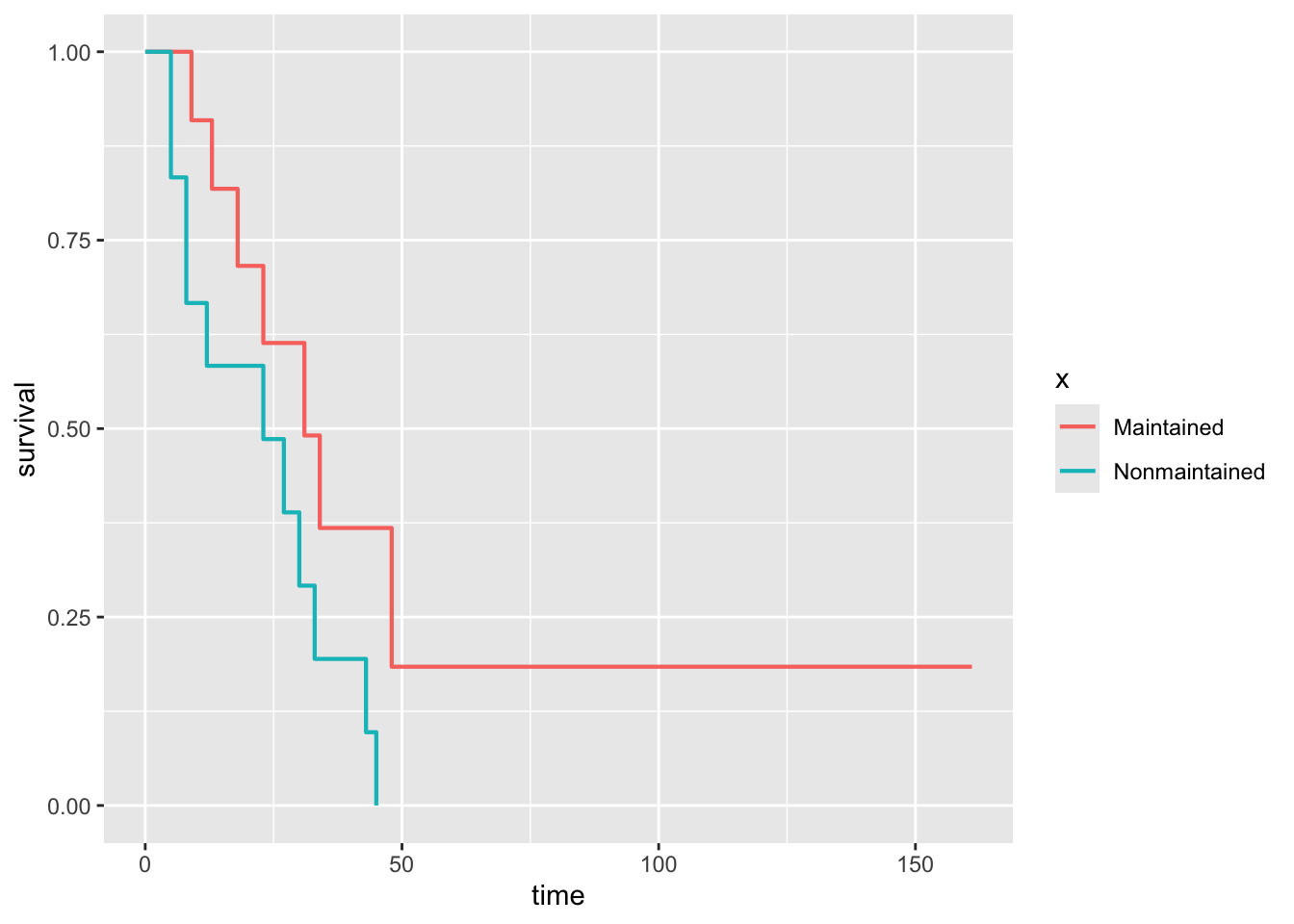
さらに、打ちきり点を表示するようにします。これにはgeom_km_ticks関数を追加します。
ggplot(data = aml,
mapping = aes(time = time, status = status, color = x)) +
geom_km() +
geom_km_ticks()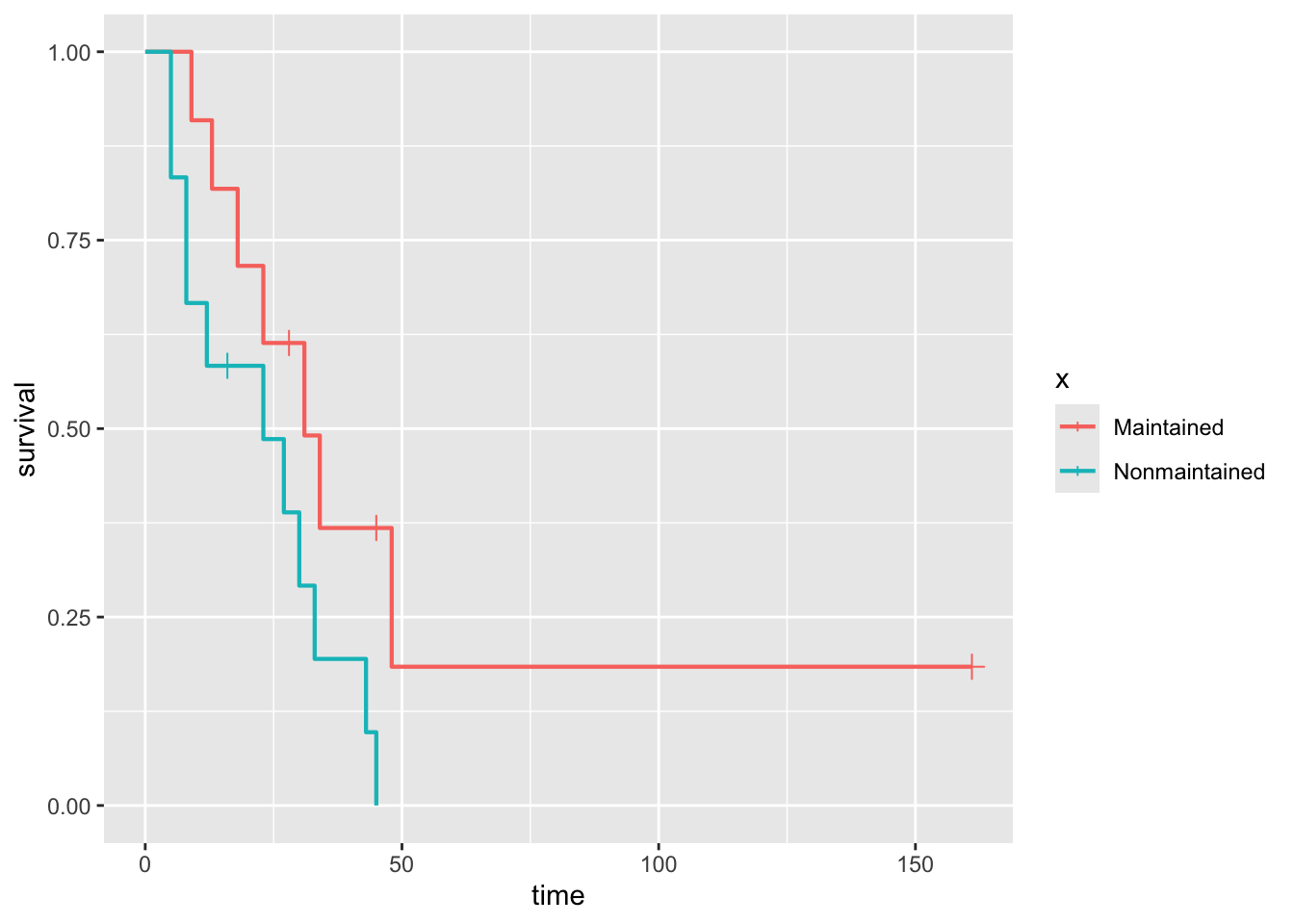
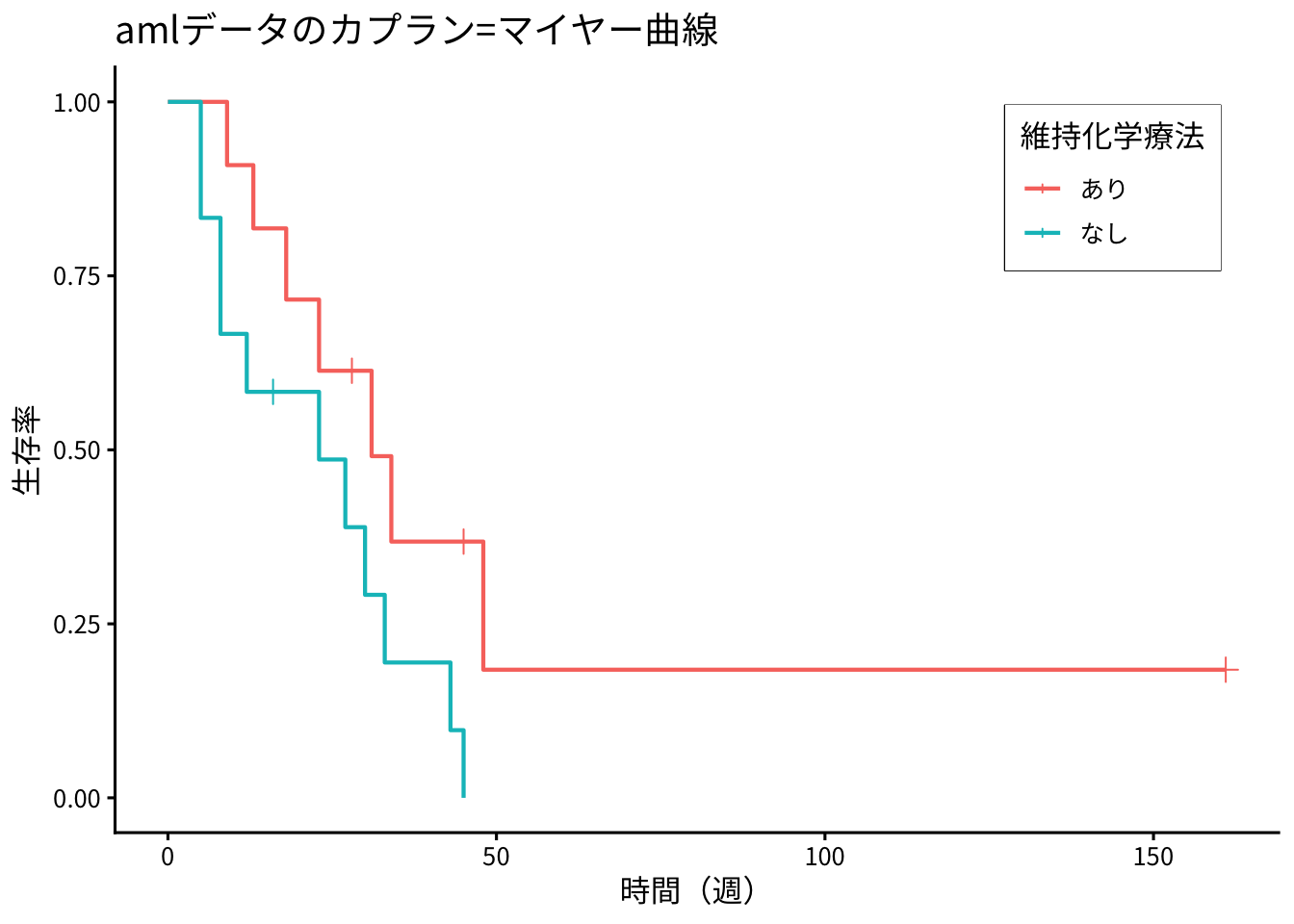
テーマや各要素の変更は、通常のggplot2そのままです。最終的には以下のようになりました。
ggplot(aml, aes(time = time, status = status, color = x)) +
geom_km() +
geom_km_ticks() +
labs(title = "amlデータのカプラン=マイヤー曲線",
x = "時間(週)", y = "生存率") +
scale_color_discrete(name = "維持化学療法",
label = c("あり", "なし")) +
theme_classic(base_family = "Noto Sans JP",
base_size = 12) +
theme(legend.position = "inside",
legend.position.inside = c(0.95, 0.95),
legend.justification = c("right", "top"),
legend.box.background = element_rect())